Bakterie
| ||
 | ||
Systematyka | ||
Domena | bakterie | |
Nazwa systematyczna | ||
| Bacteria | ||

Salmonella typhimurium – preparat mikroskopowy barwiony metodą Grama
Wyhodowane z próbek wody mikroorganizmy na pożywce agarowej
Bakterie (łac. bacteria, od gr. bakterion, pałeczka) – grupa mikroorganizmów, stanowiących osobne królestwo. Są to jednokomórkowce lub zespoły komórek o budowie prokariotycznej.
Badaniem bakterii zajmuje się bakteriologia.
Cechą charakterystyczną budowy komórek bakteryjnych jest brak otoczonych błoną organelli, takich jak jądro komórkowe czy mitochondrium, które występują u wszystkich innych organizmów żywych – grzybów, roślin, protistów i zwierząt. Wielkość komórek bakterii mieści się w zakresie od 0,2 μm dla nanobakterii do 750 μm u Thiomargarita namibiensis[1]. Mogą mieć różne kształty, np. kulisty, pałeczkowaty lub spiralny. Niektóre bakterie potrafią łączyć się ze sobą, tworząc luźne, charakterystyczne układy przestrzenne (np. pakietowce, paciorkowce, trychomy).
Bakterie występują we wszystkich biotopach. Można je spotkać w glebie, w innych organizmach i w wodzie, na lodowcach Antarktydy i wokół oceanicznych kominów hydrotermalnych. Występują także na terenach radioaktywnych, co udowodnił eksperyment, w czasie którego bakterie poddawano działaniu promieniowania jonizującego[2]. W jednym gramie gleby można znaleźć nawet 40 milionów komórek tych organizmów, a około milion w mililitrze wody słodkiej. Na Ziemi jest w przybliżeniu pięć kwintylionów (5x1030) bakterii, które stanowią znaczną część biomasy planety[3].
Dotąd nie udało się opisać wszystkich bakterii. Przyczyną jest ogromna różnorodność tej grupy organizmów, ich małe rozmiary oraz problem z przetrzymywaniem w laboratoriach – gatunki z około połowy gromad nie mogą być hodowane[4].
Pod względem sposobu odżywiania się, można je podzielić na heterotrofy i autotrofy, a także symbionty, komensale i pasożyty. Niejednokrotnie stawały się endosymbiontami.
Bakterie odgrywają ważną rolę w obiegu biogennych pierwiastków (są destruentami). Biorą udział w podtrzymywaniu wszystkich cykli biogeochemicznych (np. obiegu azotu) oraz w procesach fermentacji i gnicia. Jako symbionty żyjące w organizmach zwierząt, w tym ludzi, odpowiadają m.in. za trawienie pokarmów, umożliwiając lub przynajmniej ułatwiając w ten sposób ich odżywianie. Są producentami różnych ważnych dla funkcjonowania ekosystemu substancji, np. niektórych witamin dla konsumentów. Niektóre bakterie mogą zakłócać funkcjonowanie organizmów, powodując u nich choroby. W przemyśle i biotechnologii bakterie są niezwykle cenione, w tym przy biologicznym oczyszczaniu ścieków (jako główny element osadu czynnego) oraz przy wytwarzaniu produktów spożywczych, np. jogurtu i sera. Stosunkowo łatwo poddają się manipulacjom genetycznym, dzięki czemu mogą być wykorzystywane w przemyśle farmaceutycznym do produkcji peptydów i białek, które trudno uzyskać z innych źródeł. Modyfikowane genetycznie bakterie są producentami, między innymi, insuliny (stosowanej jako lek w terapii cukrzycy).
Spis treści
1 Historia bakteriologii
1.1 Pierwsza obserwacja bakterii
1.2 Odkrycie patogenności bakterii
1.3 Pierwszy lek przeciwbakteryjny
1.4 Wyodrębnienie archenonów
2 Morfologia
2.1 Formy morfologiczne
3 Budowa komórki
3.1 Struktury śródkomórkowe
3.2 Struktury zewnętrzne
3.3 Endospory
3.4 Znaczenie endospor u bakterii
3.5 Odporność endospor
4 Metabolizm
4.1 Podział bakterii według ich metabolizmu
4.2 Heterotrofizm u bakterii
4.3 Metabolizm fotoautotrofów
4.4 Metabolizm chemotrofów
4.5 Fermentacja u chemotrofów
4.6 Oddychanie beztlenowe litotrofów
5 Wzrost i reprodukcja
6 Genetyka
6.1 Replikony bakteryjne
6.2 Wymiana materiału genetycznego
6.3 Ochrona genomu bakterii przed wirusami
6.4 Mutacje w genomie bakterii
6.5 Koniugacja jako poziomy transfer genów
7 Ruch
7.1 Rodzaje ruchów u bakterii
7.2 Mechanizm działania wici
7.3 Rzęski politrychalne
7.4 Rzęski peritrychalne
7.5 Ruch zależny od aktyny
7.6 Rodzaje taksji u bakterii
8 Początki i wczesna ewolucja
9 Klasyfikacja i identyfikacja
9.1 Systematyka
9.2 Nomenklatura
9.3 Typy
10 Współżycie z innymi organizmami
10.1 Symbioza
10.2 Patogenność
11 Znaczenie bakterii w przemyśle
12 Przypisy
13 Bibliografia
14 Linki zewnętrzne
Historia bakteriologii |

Antoni van Leeuwenhoek był pierwszym mikrobiologiem, który przy pomocy własnoręcznie wykonanego mikroskopu zaobserwował bakterie
Pierwsza obserwacja bakterii |
Bakterie zostały po raz pierwszy zauważone w 1686 roku przez przyrodnika i przedsiębiorcę Antoniego van Leeuwenhoeka, który obserwował je używając własnoręcznie wykonanego, jednoobiektywowego mikroskopu[5]. Nazwał je „animalcules” i opublikował w serii listów do Royal Society[6].
Nazwa „bakterie” została wprowadzona znacznie później, bo w 1838 roku, od greckiego słowa baktērion (βακτηριον), czyli „pałeczka”, przez Christiana Gottfrieda Ehrenberga[7].
Odkrycie patogenności bakterii |
Louis Pasteur w 1859 wykazał, że proces fermentacji jest spowodowany przez wzrost mikroorganizmów, między innymi bakterii (oprócz nich za proces są odpowiedzialne drożdże i pleśnie, które nie są bakteriami, a grzybami). Wraz z Robertem Kochem Pasteur od samego początku był zwolennikiem teorii wywoływania chorób przez bakterie[8]. Robert Koch był pionierem w zakresie mikrobiologii medycznej. Pracował nad cholerą, gruźlicą i wąglikiem. W badaniach nad prątkami gruźlicy, Koch ostatecznie potwierdził swoją teorię dotyczącą rozwoju chorób bakteryjnych, za które przyznano mu Nagrodę Nobla w 1905[9]. Stworzył też szereg zasad zwanych postulatami Kocha, za pomocą których można stwierdzić, czy dana bakteria jest patogenem[10].
Pierwszy lek przeciwbakteryjny |
Chociaż istnienie bakterii chorobotwórczych było już pewne w XIX wieku, nie było skutecznych lekarstw do walki z nimi[11]. Paul Ehrlich opracował pierwszy lek, który nadawał się do zwalczania krętków bladych (Treponema pallidum) wywołujących kiłę, wprowadzony do obrotu w roku 1910 jako salwarsan[12][13]. Ehrlich był pionierem immunologii w wykorzystywaniu barwników do walki z bakteriami. Jego prace były podstawą do rozszerzania wiedzy o bakteriach i doprowadziły do stworzenia metody barwienia Grama, zostały też nagrodzone Nagrodą Nobla w roku 1908[14].
Wyodrębnienie archenonów |
Istotnym krokiem naprzód w badaniu bakterii było uznanie w 1977 roku przez Carla Woese, że archeony pochodzą ewolucyjnie od innych organizmów niż bakterie, z którymi nie mają większych powiązań filogenetycznych[15]. Nowa taksonomia oparta była na sekwencji 16S rRNA, co skutkowało podziałem prokariontów na dwie ewolucyjne domeny, w ramach „systemu trzech domen”[16].
Morfologia |

Schemat przedstawia porównanie wielkości bakterii na tle innych organizmów, a także cząsteczek oraz atomów
Bakterie charakteryzuje duża różnorodność kształtów i wielkości. Komórki bakteryjne są średnio około 10 razy mniejsze od komórek organizmów eukariotycznych. Osiągają od 0,5 do 5 mikrometrów wielkości. Kilka gatunków, na przykład Thiomargarita namibiensis i Epulopiscium fishelsoni, może dorastać nawet do połowy milimetra i są widoczne gołym okiem[17]. Do najmniejszych bakterii należą wszystkie z rodzaju Mycoplasma. Mają wielkość taką jak największe wirusy, osiągają maksymalnie 0,3 mikrometra[18]. Niektóre bakterie mogą być jeszcze mniejsze, ale ultramikrobakterie nie zostały na razie dokładnie zbadane[19].
Bakterie o kształcie kulistym – ziarenkowce (łac. coccus, z gr. kókkos, czyli ziarno, nasiono) – stanowią większość. Częsty jest też kształt pałeczki (łac. baculus, drążek). Niektóre bakterie o podłużnym kształcie są lekko wygięte w kształcie przecinka, dlatego nazywane są przecinkowcami (łac. vibrio). Inne mogą być w kształcie spirali – śrubowce (łac. spirilla) – lub ściśle zwiniętych sprężynek – krętki (łac. spirochetae). Niewiele gatunków ma inne, specyficzne kształty[20]. Różnorodność kształtów zależna jest od występowania i rodzaju ściany komórkowej bakterii oraz jej cytoszkieletu. Może ona wpłynąć na zdolność bakterii do zdobywania składników odżywczych, przemieszczania się w cieczach i ucieczki przed drapieżnikami oraz na możliwości czepne powierzchni komórki[21].
Wiele bakterii występuje jako pojedyncze komórki. Inne grupują się, tworząc skomplikowane formy. Bakterie z rodzaju Neisseria tworzą pary, Streptococcus przyjmują postać łańcuszków, a Staphylococcus grupują się w formy przypominające kiście winogron. Niektóre komórki są znacznie wydłużone i tworzą włókna, na przykład Actinobacteria. Bakterie Nocardia tworzą specyficzne układy, podobne do strzępków grzybni[22]. Również sinice przyjmują różne typy kolonii nitkowatych lub groniastych.
Możliwości rozwoju i kształty, jakie przybierają bakterie, najlepiej da się zauważyć przy ich hodowli na pożywkach. Skupiska bakterii, będących potomstwem jednej komórki macierzystej, nazywane są koloniami bakteryjnymi. Pojęcie to jednak nie jest tożsame z pojęciem kolonii używanym w innych dziedzinach biologii, gdzie zwykle oznacza ono skupisko osobników związanych ekologicznie i fizjologicznie, nierzadko o skomplikowanej strukturze powiązań i podziale funkcji. Kolonia bakteryjna jest po prostu wynikiem namnażania bakterii na dogodnym podłożu. Gdy mikroorganizmy trafią na odpowiednie warunki, mogą stworzyć kolonie o wielkości od kilku mikrometrów do pół metra wielkości. Duże kolonie składają się z różnorodnych grup bakterii, protistów i archeonów[22][23]. Typ wzrostu na pożywkach stałych i płynnych, oraz wygląd i zapach kolonii, często jest charakterystyczny dla danego gatunku. Niejednokrotnie kolonie bakteryjne wiążą się z podłożem, tworząc warstwy o grubości od kilku mikrometrów do ponad pół metra. Są one zwane matami bakteryjnymi lub biofilmami. Biofilmy mają, między innymi, istotne znaczenie w medycynie. Pojawiają się bowiem często w trakcie przewlekłych infekcji bakteryjnych albo na wszczepianych implantach medycznych. Bakterie występujące w formie biofilmu są przez niego chronione i z tego powodu znacznie trudniej je zabić[24].
Możliwe jest również tworzenie przez bakterie bardziej złożonych form morfologicznych. Na przykład Myxobacterie rozwijające się w środowisku ubogim w aminokwasy, lokalizują w pobliżu inne komórki i zbliżają się do nich dzięki mechanizmowi quorum sensing. Organizują się następnie w twór o długości 5000 mikrometrów (5 milimetrów), składający się w przybliżeniu z 100 tysięcy komórek bakteryjnych[25]. Poszczególne grupy komórek wykonują w nich różne złożone czynności. Na przykład około jednej dziesiątej komórek migruje w górę tej kolonii, gdzie przechodzą w stan hibernacji, przekształcając się w formy przetrwalnikowe, bardziej odporne na działanie środowiska[25]. Organizacja ta przypomina normalny organizm wielokomórkowy.
Niektóre bakterie są w stanie wytwarzać endospory, nazywane czasami przetrwalnikami, które charakteryzują się znacznym stopniem odwodnienia zawartej w nich cytoplazmy, a także grubymi i wielowarstwowymi osłonami. Endospory umożliwiają bakteriom przetrwanie w niekorzystnych warunkach, a następnie powrót do normalnych funkcji życiowych, kiedy warunki zmienią się na sprzyjające. Bakterie wytwarzające przetrwalniki należą do rodzajów Bacillus i Clostridium.
Formy morfologiczne |

Kształty kolonii bakteryjnych
ziarniaki, Micrococcus
dwoinki, Diplococcus
czworaczki[26], Tetracoccus
paciorkowce, Streptococcus
gronkowce, Staphylococcus
pakietowce, Sarcina
pałeczki, Bacterium
laseczki, Bacillus
maczugowce, Corynebacterium
przecinkowce, Vibrio
śrubowce, Spirillum
krętki, Spirochaeta
promieniowce, Actinomyces
prątki, Mycobacterium
Budowa komórki |
Wszystkie bakterie mają stosunkowo prostą budowę komórkową. Nie mają jądra komórkowego, chloroplastów, mitochondriów, aparatu Golgiego[27], które są charakterystyczne dla komórek eukariotycznych. Zamiast jądra komórkowego mają jedną dużą, kolistą i nieupakowaną cząsteczkę DNA, czyli genofor, oraz niewielkie koliste cząsteczki DNA – plazmidy.
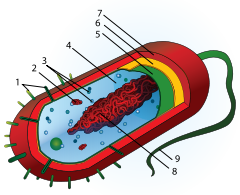
Budowa bakterii – 1. Pile, 2. Plazmid, 3. Rybosomy, 4. Cytozol, 5. Błona zewnętrzna, 6. Ściana komórkowa, 7. Otoczka, 8. Nukleoid, 9. Wić
Głównymi składnikami komórek bakteryjnych są:
- cytozol – substancja koloidalna, wypełniająca wnętrze komórki,
- nukleoid – obszar cytoplazmy, w którym znajduje się nić DNA,
- otoczka – warstwa ochronna występująca u części bakterii, zwykle zbudowana ze związków cukrowych,
- ściana komórkowa, która pełni funkcję ochronną, w jej skład wchodzi mureina,
- błona komórkowa – struktura oddzielająca wnętrze komórki od świata zewnętrznego,
- rybosomy – organelle służące do produkcji białek,
- rzęski, które są wypustkami pełniącymi funkcję ruchową, nie we wszystkich typach bakterii są obecne.
Struktury śródkomórkowe |
Komórki bakteryjne są otoczone przez błonę komórkową, która pełni rolę bariery, a także pozwala na utrzymywanie wewnątrz komórki różnych substancji, cytoplazmy i organelli. Dotyczy to np. białek, tłuszczów (są one materiałami zapasowymi większości bakterii) i innych składników odżywczych. Bakterie zaliczają się do prokariotów. Nie posiadają błony dzielącej poszczególne organelle (lub łączącej je) i w związku z tym wykształciły wiele dużych struktur śródkomórkowych. Przy pierwszej obserwacji mikroskopowej przypominały one „worki” wypełnione cytoplazmą. Później zauważono, że znajdują się tam pewne struktury, a sama cytoplazma stanowi hydroszkielet bakterii[28][29]. Stwierdzono także, że każde białko ma stałą lokalizację w cytoplazmie[30]. Bakterie posiadają mikrokompartmenty (mikroprzedziały), takie jak karboksysomy[31], które pełnią funkcję analogiczną do błony śródplazmatycznej u eukariotów (dzielą komórkę na kilka części). Są one otoczone polihedralną otoczką białkową[32]. To wielościenne organellum jest miejscem zachodzenia wielu reakcji składających się na metabolizm. Z czasem przekształciła się w siateczkę śródplazmatyczną u eukariotów[33][34].
Bakterie nie posiadają jądra komórkowego. Materiał genetyczny zlokalizowany jest w pojedynczych chromosomach oraz w plazmidach. Chromosom znajduje się w nieregularnym organellum komórkowym – nukleoidzie. Nukleoidy znajdują się w cytoplazmie[35] i zawierają oprócz chromosomów, różnorodne białka i RNA. Jedynie Planctomycetes są wyjątkiem od tej reguły. Jako jedyne mają one nukleoid otoczony błoną, która występuje także przy innych organellach i dzieli komórkę na kilka części[36]. Jak wszystkie żywe organizmy bakterie zawierają rybosomy służące do produkcji i przemian białek. Mają one jednak inną strukturę niż te występujące u archeowców i eukariotów[37].
Materiałami zapasowymi bakterii są różnorodne substancje, takie jak glikogen[38], polifosforan[39], siarka[40] lub polihydroksyalkaniany[41] (jak polihydroksymaślan). Magazynują je w ziarnkach, z których mogą być uwalniane w razie potrzeby. Fotosyntetyczne bakterie planktoniczne należące do Cyanobacteria wytwarzają pęcherzyki gazu, dzięki którym mogą regulować głębokość, na której się znajdują w toni wodnej, optymalizując w ten sposób warunki środowiska, co w cyklu 24-godzinnym, obserwujemy jako dobowe migracje pionowe[37].
Struktury zewnętrzne |

Różne typy wici występujące u bakterii. A – monotrychalne (jednorzęse) B – lofotrychalne C – amfitrychalne D – perytrychalne (okołorzęse)[42]
Podstawowe elementy struktury zewnętrznej bakterii to:
ściana komórkowa,
wić,
fimbrie i pilusy,- śluz.
Komórki bakteryjne posiadają błonę komórkową. Jest ona zwykle otoczona także dodatkowym organellum – ścianą komórkową, zbudowaną z peptydoglikanu i polisacharydów. W całość, nazywaną mureiną, łączą je peptydy zawierające D-aminokwasy[43]. Z tego powodu ściana komórkowa bakterii różni się od tych, które spotyka się u grzybów (są one zbudowane z chityny) i roślin (zbudowanych z celulozy). Bakteryjna ściana komórkowa różni się także od tych, które posiadają inne prokarioty, gdyż archeowce nie zawierają peptydoglikanu. Ściana komórkowa jest podstawową osłoną bakterii, zwiększającą ich odporność i umożliwiającą prawidłowe funkcjonowanie. Antybiotyki β-laktamowe zwalczają bakterie właśnie poprzez blokowanie syntezy peptydoglikanu, czym zaburzają budowę bakteryjnej ściany komórkowej[44].
Szczególną właściwością ścian jest ich przepuszczalność. Różnice w przepuszczalności ścian komórkowych różnych gatunków bakterii umożliwia ich identyfikację na podstawie barwienia metodą Grama. Używając tej metody rozróżniania, bakterie podzielono na bakterie Gram-dodatnie i bakterie Gram-ujemne. Zakwalifikowanie badanej bakterii do jednej z tych grup, ułatwia jej identyfikację[45].
Gram-dodatnie bakterie posiadają grubą ścianę komórkową zbudowaną z wielu warstw peptydoglikanu i kwasów teichoinowych. Z kolei bakterie Gram-ujemne mają stosunkowo cienką ścianę, również złożoną z kilku warstw peptydoglikanu, ale jest ona otoczona drugą błoną lipidową zawierającą lipopolisacharydy oraz lipoproteiny. Większość bakterii jest Gram-ujemna, tylko 2 typy, Firmicutes i Actinobacteria są Gram-dodatnie (dawniej nazywane odpowiednio bakteriami nisko Gram-dodatnimi i wysoko Gram-dodatnimi)[46]. Różnice w przepuszczalności ścian komórkowych mogą wpływać na skuteczność antybiotyków w walce z bakteriami, na przykład wankomycyna nadaje się do zwalczania bakterii Gram-dodatnich, ale jest nieskuteczna w walce z Gram-ujemnymi, takimi jak Haemophilus influenzae czy Pseudomonas aeruginosa (pałeczka ropy błękitnej)[47].
U wielu bakterii występuje warstwa „powierzchniowa” (S-layer), złożona z sztywno i ciasno ułożonych cząsteczek białka, przykrywających od zewnątrz komórkę[48]. Warstwa ta chroni bakterie przed czynnikami chemicznymi i fizycznymi, a także może stanowić wielocząsteczkową barierę, zapobiegającą przenikaniu różnych substancji ze środowiska do wnętrza komórki. S-layer ma wiele funkcji, mniej lub bardziej złożonych, jednak nie wszystkie zostały w pełni poznane. Wiadomo natomiast, że warstwa ta może decydować o zjadliwości niektórych bakterii, np. z rodzaju Campylobacter lub zawierać enzymy, pełniące rozmaite funkcje (np. u Bacillus stearothermophilus)[49].
Wić u bakterii jest sztywną strukturą białkową o średnicy 20 nanometrów i długości dochodzącej do kilku mikrometrów. Istnieją bakterie, które posiadają większą ilość wici, umożliwiających szybsze wykonywanie ruchów. Wici występujące u Prokaryota są zbudowane inaczej niż te, które posiadają organizmy zaliczane do jądrowców (Eukaryota). Wić bakterii składa się z spiralnie skręconych włókien białkowych: (flagelliny). Wici bakteryjne wprowadzane są w ruch przez występujące w błonie komórkowej bakterii białka motoryczne, które w odróżnieniu od białek motorycznych komórek eukariotycznych – nie wykorzystują cząsteczek ATP jako źródła energii, lecz czerpią ją bezpośrednio z gradientu protonowego, który występuje pomiędzy zewnętrzną i wewnętrzną stroną błony komórkowej. Białka te wprawiają w ruch obrotowy strukturę, która jest podstawą bakteryjnej wici. Gdy wici obracają się w kierunku przeciwnym do ruchu wskazówek zegara, tworzą wiązkę, która odpowiada za ukierunkowany ruch komórki. Gdy poruszają się one zgodnie z ruchem wskazówek zegara, wici zaczynają pracować skokowo i nie współpracują ze sobą, sprawiając że bakteria koziołkuje, zmieniając zarazem kierunek ruchu. Wyliczono, że bakterie posiadające wić potrafią płynąć z prędkością 25 mikrometrów na sekundę. Oznacza to, że w ciągu sekundy pokonują odległość 10 razy większą niż ich rozmiary. Gdyby ludzie poruszali się z taką szybkością, to pokonywaliby odległość 100 metrów w około 5 sekund[50].
Fimbrie natomiast, to włókna białkowe o średnicy wynoszącej 2-10 nanometrów i długości kilku mikrometrów. Są rozsiane po całej powierzchni komórki. Uważa się, że fimbrie służą do przyłączania bakterii do innych powierzchni, przez co są jednym z głównych czynników określających zjadliwość bakterii chorobotwórczych[51].
Pilusy są specyficznymi tworami nieznacznie większymi niż fimbrie. Pozwalają one na przeniesienie materiału genetycznego pomiędzy dwoma bakteriami w procesie zwanym koniugacją[52].
Bakterie wytwarzają kapsuły lub warstwy śluzu, którym się otaczają. Śluz bywa niezwykle zróżnicowany – może być zwykłą warstwą zdezorganizowanego polimeru, występującego w okolicy komórki lub złożoną otoczką stworzoną z glikokaliksu. Struktury te mają za zadanie osłaniać bakterie przed wrogami takimi jak inne bakterie, bakteriofagi lub leukocyty organizmów eukariotycznych, np. makrofagi[53]. Kapsuły i śluz mogą być również antygenami, dzięki którym bakterie mogą się rozpoznawać. Ułatwiają one również rozpoznawanie odpowiednich struktur w celu dołączenia się do nich, gdy bakterie tworzą biofilm[54].
Zgromadzenie zewnętrznych struktur komórkowych jest uzależnione od bakteryjnych systemów wydzielniczych. Białka wytwarzane we wnętrzu bakterii są wydzielane do środowiska, gdzie okładają się wokół komórki lub są wydalane całkowicie. Wiele czynników jest charakterystycznych tylko dla jednej bakterii, określają one wirulencję patogenów[55].
Endospory |

endospory Coccidioides

Mikroskopowy obraz Bacillus subtilis, widoczne endospory (zielone) wśród form wegetatywnych (czerwone)
Znaczenie endospor u bakterii |
Niektóre bakterie potrafią tworzyć struktury przetrwalnikowe zwane endosporami, które pozwalają przetrwać niekorzystne warunki. Są one wytwarzane przez bakterie Gram-dodatnie należące do rodzajów: Bacillus, Clostridium, Sporohalobacter, Anaerobacter i Heliobacterium. Charakteryzuje je niezwykła odporność[56]. W prawie wszystkich przypadkach jedna komórka wytwarza jedną endosporę, więc nie jest to proces reprodukcji, chociaż Anaerobacter potrafią wytwarzać aż siedem endospor z jednej komórki[57]. Endospora składa się z cytoplazmy wraz z DNA i rybosomami otoczonej przez nieprzepuszczalną ścianę.
Odporność endospor |
Endospory nie wykazują metabolizmu i są odporne na ekstremalne warunki fizyczne (wysokie promieniowanie UV, promieniowanie gamma, wysokie temperatury, zmiany ciśnienia i osuszanie) oraz działanie substancji chemicznych (detergenty, środki dezynfekujące)[58]. W tym stanie mikroorganizmy mogą przeczekać niekorzystne warunki, będąc gotowymi do rozwoju przez miliony lat[59][60], nawet gdy znajdą się w próżni, np. w przestrzeni kosmicznej[61]. Endospory niektórych patogenów są szczególnie groźne dla człowieka, np. Bacillus anthracis wywołujący wąglik lub Clostridium tetani wywołujący tężec. Endospory rozwijają się w żywe komórki bakteryjne wtedy, gdy dostaną się do środowiska, w którym mają ku temu dogodne warunki, np. do krwi[62].
Metabolizm |
Podział bakterii według ich metabolizmu |
W przeciwieństwie do innych grup organizmów wśród bakterii można znaleźć przedstawicieli bardzo różnych strategii metabolicznych[63]. Różnice w sposobie uzyskiwania energii i różne substancje wykorzystywane w reakcjach katabolicznych i anabolicznych zostały uwzględnione przy ich podziale systematycznym. Jednak niejednokrotnie taka klasyfikacja nie oddaje filogenezy bakterii[64]. Bakteryjne strategie metaboliczne dzieli się ze względu na trzy kryteria: źródła energii, sposoby jej uzyskiwania i substraty reakcji chemicznych. Dodatkowym kryterium jest występowanie akceptorów elektronów pozwalających na beztlenowe oddychanie[65]. Stąd jednym z kryteriów podziału jest wykorzystywanie lub niewykorzystywanie tlenu, co daje podział na bakterie tlenowe (aerobowe) i beztlenowe (anaerobowe). Podział ten bywa użyteczny przy zwalczaniu bakterii, gdyż dla wielu chorobotwórczych bakterii beztlenowych tlen jest toksyczny (anaeorobowość obligatoryjna). Wśród bakterii zdarza się też anaerobowość fakultatywna, gdy tlen nie jest konieczny do oddychania, ale nie jest też zabójczy. Niektóre bakterie tworzą kolonie, w których jedne komórki żyją w warunkach tlenowych (fotosyntezują i oddychają tlenowo), a inne (tzw. heterocysty) są od tlenu izolowane i przeprowadzają reakcje, które obecność wolnego tlenu by zaburzała (np. wiązanie azotu atmosferycznego). Jest to zjawisko typowe dla niektórych sinic (np. trzęsidłowców).
| Grupa bakterii | Źródło energii | Składniki niezbędne do uzyskania energii | Przykłady |
|---|---|---|---|
| Fotoautotrofy | Światło słoneczne | Związki organiczne lub wiązanie węgla | sinice, zielone bakterie siarkowe, Chloroflexi i bakterie purpurowe |
| Litotrofy | Związki nieorganiczne | Związki organiczne lub wiązanie węgla | Thermodesulfobacteria, Hydrogenophilaceae, i Nitrospirae |
| Organotrofy | Związki organiczne | Związki organiczne (chemoheterotrofy) lub wiązanie węgla (chemoautotrofy) | Bacillus, Clostridium i Enterobacteriaceae |
Heterotrofizm u bakterii |
Metabolizm węgla może być u bakterii heterotroficzny, gdzie związki organiczne zostają wykorzystane do produkcji energii. Wiele bakterii heterotroficznych jest pasożytami innych organizmów. Znane są również bakterie autotroficzne, zdobywające węgiel z dwutlenku węgla. Do typowych bakterii autotroficznych zalicza się cyjanobakterie (sinice), zielone bakterie siarkowe, niektóre bakterie purpurowe, ale też wiele chemolitotrofów, takich jak bakterie nitryfikacyjne i bakterie siarkowe[66].
Metabolizm fotoautotrofów |
Innym kryterium podziału bakterii jest sposób produkcji użytecznej biologicznie energii – fotoautotrofy, używające światła do fotosyntezy oraz chemoautotrofy, które potrzebują substancji chemicznych do uzyskiwania energii w trakcie ich utleniania. Utleniaczem może być tlen, ale w warunkach beztlenowych także inne substancje.
Poza tym bakterie dzieli się na litotrofy, które używają związków mineralnych jako donorów elektronów oraz organotrofy, które jako donorów używają związków organicznych.
Metabolizm chemotrofów |
Organizmy chemotroficzne używają do wytwarzania energii, w procesach oddychania i innych reakcjach (np. biosyntezie), określonych substancji chemicznych. Fototrofy używają ich tylko do biosyntezy, natomiast energię czerpią ze światła. Organizmy oddychające, jako źródło energii wykorzystują związki chemiczne, pobierając elektrony od zredukowanego substratu i przenosząc je do akceptorów (reakcja redoks). Energia ta może zostać użyta do wytworzenia ATP i w postaci tego nośnika – do kolejnych reakcji metabolicznych. U organizmów tlenowych akceptorem elektronów (tj. utleniaczem) jest tlen. U bakterii beztlenowych są to inne substancje chemiczne, np. azotany, siarczany albo dwutlenek węgla. Reakcje te są podstawą wielu ważnych ekologicznie procesów, takich jak denitryfikacja, redukcja siarczanów i octanogeneza.
Fermentacja u chemotrofów |
Innym sposobem życia chemotrofów, występującym w przypadku braku wyżej wymienionych akceptorów elektronów jest wykorzystywanie fermentacji, gdzie elektrony zredukowanego substratu są przenoszone do utlenionych nośników pośrednich, aby wytworzyć zredukowane produkty fermentacji (np. etanol). Fermentacja jest możliwa, ponieważ energia zawarta w substratach jest większa niż energia zawarta w produktach reakcji, dzięki czemu mogą one zsyntezować ATP i wykorzystać ten typ metabolizmu[67][68].
Ten proces ma także wielkie znaczenie w biologicznej reakcji na zanieczyszczenie. Bakterie siarkowe charakteryzuje produkcja niezwykle toksycznych substancji na bazie rtęci (metylortęci i dimetylortęci). Anaerobowe bakterie korzystają z fermentacji pozwalającej wytworzyć energię i siłę redukcyjną, przy czym dochodzi do ubocznego wytwarzania metabolitów (takich jak etanol, co ma znaczenie np. w piwowarstwie)[69]. Fakultatywne anaeroby mogą przechodzić między fermentacją a wykorzystywaniem innych różnorodnych akceptorów elektronów, w zależności od warunków środowiska.
Oddychanie beztlenowe litotrofów |
Litotrofy mogą używać związków nieorganicznych jako źródła energii. Donorami elektronów w tym przypadku są substancje takie jak wodór, tlenek węgla, amoniak (proces nitryfikacji), monotlenek żelaza (tlenek żelaza(II)), zredukowane jony żelaza i kilka (również zredukowanych) związków siarki. Metan może także posłużyć bakteriom metanotroficznym do anabolizmu węgla i jako źródło elektronów[69]. Zarówno aerobowe fototrofy, jak i chemolitotrofy, używają tlenu jako ostatecznego akceptora elektronów, jednak w sytuacji niedoboru tlenu przechodzą one z powrotem na wykorzystywanie w tym celu związków nieorganicznych. Litotrofy w większości są autotroficzne, natomiast organotrofy – heterotroficzne.
Pewne bakterie potrafią wiązać azot atmosferyczny w procesie diazotrofii. Są to bakterie azotowe, czasem żyjące w symbiozie z roślinami jako bakterie brodawkowe. Ten szlak metaboliczny mający duże znaczenie dla środowiska (geochemiczny obieg azotu) można znaleźć u bakterii przyjmujących niemal wszystkie strategie metaboliczne, zatem niektóre bakterie potrafią wiązać zarówno atmosferyczny azot, jak i węgiel (w procesie fotosyntezy), nie jest jednak uniwersalny[70].
Wzrost i reprodukcja |
Wzrost komórek bakteryjnych, podobnie jak wszystkich innych komórek, polega na stopniowym zwiększaniu objętości, poprzez produkcję nowych białek i innych elementów strukturalnych, oraz gromadzenie substancji pochodzących ze środowiska. Bakterie rosną, aż osiągną pewną wielkość, po której następuje ich podział na dwie bakterie pochodne. Podział ten nosi nazwę podziału komórki i jest jednym ze sposobów rozmnażania bezpłciowego[71]. Przy odpowiednich warunkach bakterie mogą dzielić się co około 9,8 minuty[72]. Komórki potomne są identyczne z rodzicielską.
Niektóre bakterie posiadają bardziej złożoną budowę i struktury ułatwiające rozmnażanie, np. Myxobacteria i Streptomyces. Bakterie te wytwarzają strzępki na swojej powierzchni, które następnie dojrzewają i odrywają się od komórki macierzystej.
U niektórych typów bakterii mogą występować procesy płciowe, lecz nie mają one żadnego związku z reprodukcją.

Burkholderia pseudomallei po 96 godzinach na pożywce w temperaturze 37 °C
Burkholderia pseudomallei po 72 godzinach na podłożu mikrobiologicznym
W laboratoriach w celu obserwacji i hodowli, bakterie umieszcza się na odpowiednich stałych lub ciekłych podłożach hodowlanych (mikrobiologicznych). Stałe, takie jak pożywka agarowa (której głównym wypełniaczem jest pozyskiwany z glonów agar-agar) są używane w celu izolacji odpowiednich kultur bakteryjnych. Z płynnych korzysta się najczęściej, gdy należy wykonać pomiar wzrostu dużych ilości bakterii lub szacować ich ilość w badanym materiale. Komórka w pożywce ma zapewnione wszystkie niezbędne jej do życia składniki. Dodatkowo mogą one bardzo łatwo rozprzestrzeniać w całym podłożu. Trudno jednak wyizolować pojedyncze komórki, ponieważ bakterie mnożą się bardzo szybko i w krótkim czasie wypełniają prawie całą, dostępną im powierzchnię. Mieszaniny pożywek z dodatkowymi substancjami (np. antybiotykami) pozwalają na sprawdzenie właściwości bakterii, a także na przypisanie ich do danego podgatunku[73].
W większości laboratoriów do hodowli bakterii przeznacza się odpowiednie składniki odżywcze, w celu osiągnięcia jak największej ilości bakterii w krótkim czasie. Faza intensywnego wzrostu populacji bakterii na danej pożywce jest czasem określana w przybliżeniu jako faza wzrostu wykładniczego.
Jednakże w naturalnych warunkach, w których może braknąć pożywienia, bakterie nie mogą rozmnażać się bez przerwy. Z tego powodu opracowały one odpowiednią strategię rozrodczą. Gdy nastąpi nagły wzrost ilości pożywienia bakterie mogą gwałtownie się rozmnażać, podobnie do glonów, które w takich warunkach (zwykle latem) tworzą zakwity[74]. Przez dobór odpowiednich składników podłoża można również prowadzić hodowle różnicowe służące do identyfikacji mikroorganizmów.
Wzrost bakterii można podzielić na trzy etapy. Kiedy środowisko zmienia się, np. zwiększa się ilość pożywienia, albo bakterie dostaną się do nowego miejsca, muszą odpowiednio przystosować się do nowych warunków życia. Zmiany te wpływają także na ich rozwój. Pierwszym stadium rozwoju bakterii jest tzw. faza pierwotnego zahamowania. W tym czasie rozwijają się one bardzo wolno przygotowując się do zwiększenia ilości pożywienia w ich pobliżu, dzięki któremu będą mogły szybko urosnąć. Stadium charakteryzuje wysoki poziom biosyntezy niezbędny do wyprodukowania dużych ilości białka[75]. Drugim stadium jest faza logarytmicznego wzrostu, znane też jako stadium wykładnicze. Charakteryzuje je niezwykle szybki wzrost bakterii. Tempo wzrastania komórek jest oznaczane jako tempo wzrostu (k), a czas jaki jest potrzebny do podziału nazywa się czasem pokolenia (g). Podczas tej fazy zwiększa się metabolizowanie pożywienia, aż go zabraknie, co spowoduje zatrzymanie procesu i przejście do fazy trzeciej. Jest to stadium, które stanowi moment przejścia pomiędzy poprzednimi. Bakterie powoli stabilizują swoje procesy życiowe, część z nich obumiera. Te które przetrwają wracają do fazy zahamowania[76].

Wzrastająca kolonia Escherichia coli[77]
Bakterie wypracowały wiele różnych mechanizmów ułatwiających im przetrwanie. Jedną z możliwości Streptomyces jest produkcja antybiotyków (streptomycyna, neomycyna) w celu likwidacji innych drobnoustrojów znajdujących się w pobliżu. Sama bakteria jest na nie odporna[78]. Oprócz tego część bakterii wytwarza bakteriocyny, które doprowadzają do bakteriostazy, czyli zahamowania wzrostu wrażliwych na nie gatunków. Dodatkowo wiele bakterii przystosowało się do życia w środowisku tworząc kolonie (np. biofilmy) w miejscu o zwiększonej ilości pożywienia. Dzięki temu mogą lepiej pozyskiwać substancje odżywcze ze środowiska[79]. Te sposoby uzyskiwania lepszych warunków do rozwoju są niezwykle cenne, gdyż zapewniają minimalizację kosztów wynikających z konkurencji i częściowe bezpieczeństwo[80].
Genetyka |
Replikony bakteryjne |
Bakterie posiadają zwykle pojedynczy chromosom, którego wielkość wynosi zaledwie od około 160 000 par zasad u endosymbiotycznych Candidatus Carsonella ruddii[81] do 13,033,779 par u Sorangium cellulosum[82]. Krętki z rodzaju Borrelia są jedynym wyjątkiem, gdyż mają pojedynczy chromosom liniowy. Należy tu Borrelia burgdorferi, który wywołuje boreliozę, zwaną także krętkowicą kleszczową oraz chorobą z Lyme[83]. Bakteryjne geny zwykle są umieszczone na pojedynczym odcinku DNA i chociaż u bakterii zdarzają się także różne typy intronów, to są one o wiele rzadsze niż u eukariontów[84]. Bakterie mogą także posiadać plazmidy, które są cząsteczkami DNA replikującymi się samodzielnie, niezależnie od chromosomu bakteryjnego. Plazmidy, w przeciwieństwie do chromosomów bakteryjnych, nie niosą genów metabolizmu podstawowego, nie są więc bezwzględnie niezbędne do życia komórki bakteryjnej. Mogą jednak nieść geny odpowiadające np. za oporność na antybiotyki, oraz wirulencję bakterii.
Wymiana materiału genetycznego |
Bakterie potrafią także wymieniać się genami między sobą. Zjawisko to może nastąpić na trzy różne sposoby. Po pierwsze bakterie mogą pobrać dany gen z środowiska w procesie nazywanym transformacją. Drugą metodą jest przeniesienie genów w procesie transdukcji, kiedy bakteriofag wprowadza swoje geny do chromosomu. Trzecią możliwość stanowi koniugacja bakteryjna, gdzie DNA jest przenoszone poprzez bezpośredni kontakt komórek bakterii.
Ochrona genomu bakterii przed wirusami |
Jednym z typów bakteryjnego genomu jest połączenie genów bakterii z materiałem genetycznym bakteriofagów. Wiele bakterii jest atakowanych przez te wirusy, które „wstrzykują” do ich chromosomów własny materiał genetyczny. Bakteriofagowe geny mają wielki wpływ na fenotyp bakterii. Dzięki temu na drodze ewolucji niegroźne bakterie zamieniały się w niebezpieczne dla życia innych organizmów, takie jak Escherichia coli O157:H7 czy Clostridium botulinum po przejęciu niesionych przez faga genów pewnej toksyny[85]. Bakterie potrafią jednak bronić się przed zainfekowaniem przez fagi. Służy im do tego specjalny system obronny nazywany systemem ograniczenia modyfikacji, który potrafi degradować obce DNA i rozbijać je na pojedyncze nukleotydy[86]. System ten używa także odpowiednich kolejności CRISPR, dzięki któremu może zapamiętywać szczególne cechy danego bakteriofaga i w późniejszym kontakcie z nim znacznie lepiej go zwalczać, poprzez blokowanie syntezy materiału genetycznego faga i bakterii oraz dalszemu uniemożliwieniu przeprowadzenia intenferencji RNA[87][88]. CRISPR stanowi więc odporność nabytą bakterii na infekcje wirusową.
Mutacje w genomie bakterii |
Bakterie jako organizmy rozmnażające się bezpłciowo dziedziczą identyczny materiał genetyczny od ich rodziców (można nawet powiedzieć, że są ich klonami). Mimo to każda bakteria kształtuje swój własny fenotyp, co jest spowodowane zmianami w DNA. Poza tym wśród bakterii można sztucznie spowodować zmianę. Naturalnie może ona powstać na skutek mutacji oraz rekombinacji genetycznej. Mutacje powstają na skutek błędów w czasie tworzenia repliki DNA lub jako skutek oddziaływania mutagenu. Szansa na powstanie i czas potrzebny do zajścia mutacji są różne w obrębie każdego gatunku, a nawet tej samej bakterii[89]. Genetyczne zmiany u bakterii są powodowane mutacjami przy replikacji genów albo na skutek różnorodnych „nacisków” ze strony człowieka, gdy na wybrany gen oddziałuje się licznymi mutagenami, prowadzi to do zakłócenia procesów wewnętrznych i w konsekwencji do mutacji[90].
Koniugacja jako poziomy transfer genów |
Podczas koniugacji jedna komórka („dawca”) wytwarza rurkowate cytoplazmatyczne wyrostki, tzw. pilusy, umożliwiające kontakt między komórkami bakterii. Po wymianie cytoplazmy wraz z materiałem genetycznym (plazmidami) komórki rozdzielają się. Proces ten ma różne odmiany. Wszystkie sposoby wymiany materiału genetycznego nazywają się poziomym transferem genów. Bakterie mogą wykorzystywać wszystkie te metody w naturalnym środowisku[91]. Transfer genowy jest szczególnie cenny przy wytwarzaniu oporności na antybiotyki, gdyż umożliwia bakterii odpornej na działanie antybiotyku uodparnianie innych, poprzez przekazywanie im genów warunkujących odporność, przez co może doprowadzić do uodpornienia całej populacji[92]. Z tego powodu poziomy transfer genów może być z medycznego punktu widzenia niezwykle groźny, gdy zachodzi wśród bakterii chorobotwórczych. Odkrycie poziomego transferu genów (transformacji u dwoinki zapalenia płuc) przez Fredericka Griffitha przyczyniło się do rozwoju genetyki molekularnej i później pozwoliło wyjaśnić rolę DNA.
Ruch |

Schemat wici Gram-ujemnej bakterii.
Rodzaje ruchów u bakterii |
Część bakterii posiada zdolność do aktywnego ruchu. Poruszanie może odbywać się za pomocą wici, ruchu ślizgowego, ruchu wirowego lub zmian wyporności[93]. W przypadku ruchu wirowego bakteria używa IV typu pilusów, korzystając z nich jak z haka. Mocuje się nimi w podłożu i podciąga się ze stosunkowo dużą siłą (>80 pikoniutonów)[94]. Mikroby mogą poruszać się z dużą prędkością, na przykład przecinkowiec cholery (Vibrio cholerae) należący do najbardziej ruchliwych bakterii osiąga prędkość 200 μm/s, czyli w ciągu minuty jest w stanie pokonać odległość większą niż 1 cm. Ovobacter propellens potrafi poruszać się z prędkością nawet 1 mm/s[95].
Mechanizm działania wici |
Bakterie charakteryzuje niezwykła różnorodność wici. Może ona być pojedyncza i długa, podwójna (jedna z przodu, a druga z tyłu) lub wiązka wici na części lub całej komórce bakteryjnej. Wić należy do najlepszych środków napędu wykorzystywanych przez większość organizmów. Tworzy ją 20 białek, ale do jej kontroli i przymocowania bakteria używa ich 30[93]. Wić to obracająca się struktura zewnątrzkomórkowa wprawiana w ruch przez kinetosom, który napędza gradient H+ wytwarzany przez pompę protonową.
Rzęski politrychalne |
Wiele bakterii (np. Pałeczka okrężnicy (E. coli)) posiada możliwość wykonywania kilku różnych ruchów: w przód (pływanie), w dół lub w górę. Możliwości wykonywania kilku ruchów sprawiają, że mogą one poruszać się trójwymiarowo w błądzeniu losowym[96].
Rzęski peritrychalne |
Unikatowy rodzaj wici posiadają krętki (Spirochateae), u których są one umieszczone pomiędzy dwoma błonami w przestrzeni międzyplazmatycznej. Ich ciało ma charakterystyczny kształt linii śrubowej, wijąc się, gdy bakteria porusza się[93].
Ruch zależny od aktyny |
Niektóre Listeria i Shigella potrafią wykorzystywać cytoszkielet wewnątrz komórki gospodarza. Wytwarzają strukturę przypominającą ogon lub wić, która ułatwia im pokonywanie dużych odcinków w ciele żywiciela[97].
Rodzaje taksji u bakterii |
Ruchliwe bakterie reagują na bodźce, wykonując ruchy do przodu lub do tyłu. Bakterie poruszają się dzięki różnym taksjom (chemotaksja, fototaksja, magnetotaksja i in.)[98][99]. Specyficzna pod tym względem jest grupa Myxobacteria, wśród której bakterie poruszają się grupowo, tworząc owocniki[27]. Ruchy myksobakterii można zaobserwować tylko na stałych pożywkach, w przeciwieństwie do E. coli, która wykonuje ruchy wyłącznie w płynach.
U różnych bakterii występują następujące rodzaje taksji:
- geotaksje, jako efekt zmian metabolizmu, pionowe przemieszczanie się w toni wodnej;
fototaksje, wywołane reakcjami związanymi z wyszukiwaniem optymalnych warunków świetlnych;
chemotaksje, czyli ruchy od lub do substancji chemicznych obecnych w środowisku; pozwala wyszukać pokarm lub rozpoznać obecność innych komórek; bodźce chemiczne odbierane są tu za pomocą białek receptorowych znajdujących się w błonie komórkowej;- aerotaksje; ruch ku miejscom o optymalnym stężeniu tlenu (pewien rodzaj chemotaksji);
- magnetotaksje; są to ruchy wzdłuż pola linii pola magnetycznego, tak by bakteria żyjąca w mule wędrowała w głąb osadu (ku warunkom beztlenowym); umożliwiają je specjalne organelle – magnetosomy zwierające tlenki żelaza, umieszczone w pobliżu nasady rzęsek[26].
Początki i wczesna ewolucja |
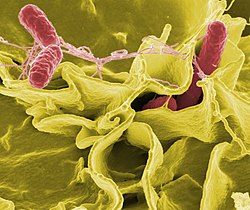
Podkolorowany obraz mikroskopowy bakterii Salmonella typhimurium (czerwone) na ludzkich komórkach (żółte)
Przodkami współczesnych bakterii były jednokomórkowe mikroorganizmy, które pojawiły się na Ziemi jako pierwsze formy życia około 4 miliardów lat temu. Przez około 3 miliardy lat wszystkie organizmy były mikroskopijne, a bakterie i archeowce były dominującymi formami życia[100][101] (do pojawienia się pierwszych bezkręgowców i roślin). Istnieją skamieniałości bakterii, takie jak stromatolity, jednak brak znaczących różnic morfologicznych pomiędzy nimi a współczesnymi bakteriami, uniemożliwia ocenianie na ich podstawie wieku różnych gatunków, a także traktowania ich jako skamieniałości przewodnich. Natomiast analiza sekwencji genowych pozwala określić niektóre funkcje dawnych komórek bakteryjnych. Wykazano, że te pierwsze komórki bakteryjne różniły się od pierwszych linii organizmów eukariotycznych i archeowców[102]. Ostatnim wspólnym przodkiem organizmów bakteryjnych i Archea był organizm hipertermofilny, który występował najprawdopodobniej około 2,5 miliardów do 3,2 miliardów lat temu[103][104]. Istniejące dziś rozbieżności między domenami Eubacteria, Eukaryota i Archaea są wynikiem różnicowania się podczas długiej ewolucji. Uważa się, że dzisiejsze organizmy eukariotyczne powstały poprzez wejście w symbiozę z ówczesnymi komórkami bakteryjnymi[105][106].
Pomiędzy komórkami prokariotycznymi a eukariotycznymi istnieją liczne różnice. U prokariontów brak na przykład występujących w niemal wszystkich komórkach eukariotycznych organelli komórkowych takich jak jądro komórkowe czy mitochondria (lub hydrogenosomy). W XX w. sugerowano, że odpowiednikiem mitochondriów lub hydrogenosomów mogą być mezosomy, które ostatecznie jednak okazały się artefaktem[107]. Mitochondria wykazują pewne podobieństwa w swojej budowie do pewnych bakterii, co daje podstawy do stwierdzenia, że bakterie (zapewne pierwotne proteobakterie) na drodze endosymbiozy zostały wchłonięte przez eukarionty i przekształciły się w mitochondria. Niektóre bakterie endosymbiotyczne (sinice) zredukowały się wewnątrz komórek eukariotycznych tworząc chloroplasty i prowadząc do powstania glonów i roślin. Istnieje wiele grup glonów, w przypadku których wykazano znaczne podobieństwa, w tym genetyczne, ich chloroplastów i bakterii, co pozwala twierdzić, że ich ewolucja zaczęła się od komórek bakterii[108][109].
Klasyfikacja i identyfikacja |

Drzewo filogenetyczne pokazujące wspólne pochodzenie organizmów wszystkich trzech domen. Bakterie są oznaczone kolorem niebieskim, eukariota czerwonym, a archaea – zielonym. Względne umiejscowienie niektórych typów ukazane jest dookoła drzewa.
Klasyfikacja biologiczna bakterii stara się opisać dokładnie każdy ich gatunek oraz zgrupować je według pokrewieństwa. Bakterie można sklasyfikować na podstawie różnic w ich budowie (np. występowania ściany komórkowej), typie metabolizmu albo na różnicach w organellach komórkowych i DNA, np. po występowaniu kwasów tłuszczowych, pigmentu, antygenów i benzochinonu[73]. Mimo to używanie tych różnic jako kryteriów nie było najlepszym wyjściem, gdyż nie dawało pewności, czy dane bakterie należą do jednego gatunku, ale są dosyć zróżnicowanymi odmianami, czy też należą do dwóch oddzielnych taksonów. Główną przyczyną tych niepewności był fakt, że nawet przy użyciu zaawansowanego sprzętu bakterie są do siebie wizualnie bardzo podobne i nie posiadają wyróżniających je struktur oraz istnienie różnych postaci poziomego transferu genów między niespokrewnionymi ze sobą gatunkami[110]. Dzięki temu transferowi nawet niezwykle spokrewnione ze sobą bakterie mogą przeprowadzać inny metabolizm i mieć inne organelle. Dla zwiększenia precyzji taksonomicznej w systematyce bakterii wykorzystuje się liczne osiągnięcia biologii molekularnej, która używa różnorodnych technik do badania DNA, np. sprawdzanie ilości cytozyny i guaniny w hybrydyzacji genomu oraz badaniu materiału nieulegającego transferowi, tj. rRNA[111]. Najnowsze dane dotyczące systematyki bakterii podaje IJSEM (International Journal of Systematic Bacteriology) oraz Bergey’s Manual Trust.
Słowo „bakteria” było używane do wszystkich mikroskopijnych organizmów. Z czasem biolodzy molekularni i systematycy doszli jednak do wniosku, iż było to błędne założenie. Wszystkie mikroorganizmy podzielono na dwie grupy; Eubacteria i Archaebacteria, które teraz nazywa się w skrócie Bacteria i Archaea, które rozwinęły się niezależnie ze wspólnego przodka[16]. Według niektórych naukowców to archeowce i eukarionty są ze sobą bardziej powiązane niż obie te grupy z bakteriami. Poza tym obie te grupy wraz z bakteriami są podstawą systemu trzech domen, który jest aktualnie najczęściej używany przy tematyce mikrobiologicznej[112]. Mimo to problemy z obserwacją bakterii oraz przy omawianiu ewolucji sprawiają, że w taksonomii bakterii i archeowców bardzo często pojawiają się zmiany[4]. Przykładowo, niektórzy biolodzy argumentują, że archeany i eukarionty wyewoluowały z bakterii Gram-dodatnich[113].
Prawidłowa identyfikacja bakterii w laboratorium ma szczególne znaczenie w medycynie. Dzięki niej można mieć pewność co do patogenu, z jakim ma się do czynienia. Choroby były jedną z przyczyn rozwoju bakteriologii i systematyki bakterii, ponieważ lekarze nie wiedzieli z czym i jak mają walczyć.
Barwienie metodą Grama, opracowane przez Hansa Christiana Grama w roku 1884, było odkryciem przełomowym w dziedzinie mikrobiologii (zwłaszcza lekarskiej). Jego metoda bazowała na charakterystyce budowy ściany komórkowej bakterii[114]. Grube warstwy peptydoglikanu u bakterii Gram-dodatnich są barwione na kolor purpurowy, podczas gdy Gram-ujemne o znacznie cieńszej ścianie komórkowej są barwione na różowo. Dzięki informacjom uzyskanym z wyników barwienia oraz po cechach morfologicznych organizmów można je zakwalifikować do ziarenkowców, laseczek, pałeczek. Mimo to istnieją patogeny, których nie można sklasyfikować na podstawie testu Grama, np. prątki lub Nocardia, do wykrywania których wykorzystuje się barwienie kwasowe i barwienie metodą Ziehla-Neelsena[115]. Istnieją jednak bakterie, które można zidentyfikować tylko na podstawie obserwacji ich wzrostu w pożywkach lub innych technik, np. serologicznych.
Kultury bakteryjne oraz techniki ich pozyskiwania są różne dla różnych bakterii. W plwocinie można znaleźć bakterie, które wywołują np. zapalenie płuc. Do innych stosuje się badanie kału lub moczu. Bakterie takie jak salmonella można zidentyfikować na podstawie reakcji, w jakie wchodzą z innymi bakteriami na pożywkach. Substancje i płyny, takie jak krew lub płyn mózgowo-rdzeniowy, które w normalnych sytuacjach są sterylne, w przypadku infekcji zapełniają się bakteriami. W przypadku błędnie pobranego materiału wyhodowanie bakterii nie musi oznaczać infekcji. Dodatkowo wszelkie bakterie jakie w nich się znajdują można przenieść na pożywki w celu ich hodowli i identyfikacji[73][116].
Jako metodę klasyfikacji bakterii coraz częściej używa się badań i eksperymentów molekularnych. Dzięki reakcji łańcuchowej polimerazy można szybko i łatwo (w warunkach laboratoryjnych) sprawdzić sekwencje genomu bakterii i porównać je z różnymi szczepami[117]. Te metody pozwalają także na wykrywanie „żywych, ale nie rozmnażających się” bakterii, które przeprowadzają metabolizm, ale nie są w stanie dokonywać podziałów[118]. Jednak mimo używania najlepszych znanych ludzkości metod badawczych nie można dokładnie stwierdzić ile gatunków bakterii istnieje na Ziemi ani ile szczepów zawiera dany gatunek. Ludzie znają mniej niż 9000 gatunków bakterii (łącznie z sinicami)[119], a oszacowania mówią o istnieniu od 107 do 109 gatunków bakterii, przy czym prawdopodobnie istnieje ich znacznie więcej[120][121].
Systematyka |
Ze względów historycznych termin „bakterie” bywa rozumiany nieprecyzyjnie. Tradycyjnie (mniej więcej do połowy XX w.) obejmował wszystkie prokarionty z wyjątkiem sinic, zaliczanych do glonów. Typ pierwszy określano jako Bacteriophyta (rozprątki) z jedną klasą – Bacteria (bakterie), typ drugi – jako Cyanophyta (glony niebieskozielone) z jedną klasą Cyanophyceae (sinice). Obydwa typy zaliczano do roślin w ówczesnym rozumieniu tego pojęcia, czasem łącząc je w grupę Schizophyta (rozprątki) lub Akaryobionta (bezjądrowe)[122]. Już jednak w systemie Ernsta Haeckla grupa Moneres została włączona do odrębnego od roślin i zwierząt królestwa Protista. System ten jednak nie zyskał powszechnej akceptacji, a bakterie traktowane były jak specyficzne rośliny zarodnikowe[123], a mikrobiologia (zwłaszcza w zakresie niezwiązanym bezpośrednio z medycyną) jak gałąź botaniki.
Przez długi czas uważano, że główna linia podziału bezjądrowców znajduje się między typowymi bakteriami (nazywanymi wówczas po prostu „bakteriami”) a sinicami. Dopiero w drugiej połowie XX w. odkryto, że istotniejsze różnice występują między typowymi bakteriami, łącznie z sinicami, a tzw. „archebakteriami”. Nazwa „archebakterie” sugeruje, że są one najstarszą ewolucyjnie zachowaną gałęzią bakterii, gdyż odkryto je w ekstremalnych środowiskach, przypominających pod pewnymi względami warunki pierwotnej Ziemi. Obecnie uważa się inaczej, ale nazwa „archeany” (bez członu „bakterie”) pozostała w użyciu.
Według „systematyki pięciu królestw” wszystkie prokarionty zgrupowano w jedno królestwo Monera z dwoma podkrólestwami: Eubacteria (czyli „bakterie właściwe”) i Archaea (archeany), a sinice zaliczono do tych pierwszych jako niższy takson.
Dokładniejsze badania na poziomie molekularnym zasugerowały, że z ewolucyjnego punktu widzenia archeany są równie odległe od reszty prokariontów, jak od eukariontów, a pod pewnymi względami nawet bliższe tym ostatnim. Spowodowało to zaproponowanie „systematyki trzech domen”, według której „bakterie właściwe” stanowią jedną z domen, obok archeanów i eukariontów. W takim ujęciu słowo „bakteria” powinno odnosić się do podkrólestwa Eubacteria, równoważnego z domeną Bacteria. Należy jednak zaznaczyć, że nie wszyscy naukowcy zgadzają się z taką interpretacją, wskazują na błędy w interpretacji danych molekularnych i uważają, że termin „bakterie” winien być używany także wobec „archeanów”.
Nomenklatura |
Zasady nazewnictwa taksonów bakteryjnych do 1975 r. były takie jak w przypadku roślin i grzybów. Od tego roku obowiązuje odrębny, Międzynarodowy Kodeks Nomenklatury Bakterii (ang. The International Code of Nomenclature of Bacteria, ICNB). Wszystkie uznawane za poprawne nazwy naukowe bakterii, po weryfikacji ich poprawności wobec zasad określonych w kodeksie, umieszczane są na liście „Approved Lists of Bacterial Names” i publikowane w czasopiśmie „International Journal of Systematic and Evolutionary Microbiology”[124]. Celem kodyfikacji nomenklatury naukowej bakterii jest stworzenie stabilnego systemu nazewnictwa, pozwalającego na unikanie nieporozumień i dublowanie nazw. Zgodnie z kodeksem (reguła 5b ICNB) bakterie dzielone są na 5 kategorii: gatunki (species), rodzaje (genus), rodziny (familia), rzędy (ordo) i klasy (classis). Podział może być uszczegóławiany poprzez dodawanie kategorii pomocniczych: podgatunek, podrodzina, plemię i podplemię, podrząd i podklasa. Odmiana (varietas) w nomenklaturze bakteryjnej jest synonimem podgatunku. Skutkiem wywodzenia się nomenklatury bakteryjnej z botanicznej jest wiele podobieństw, np. identyczne typowe końcówki nazw dla taksonów odpowiednich rang systematycznych jak u protistów roślinopodobnych zgodnie z Międzynarodowym Kodeksie Nomenklatury Botanicznej, zapis kursywą nazw naukowych różnych rang systematycznych[125].
Typy |

Bakterie żelazowe w rzece w Szkocji
Niezależnie od przyjętego systemu klasyfikacji domen i królestw wśród bakterii wyróżnia się następujące typy[126][127]:
Acidobacteria Cavalier-Smith 2002 – kwasolubne bakterie glebowe
Actinobacteria (Stackebrandt i inni, 1997) Cavalier-Smith 2002 – Gram-dodatnie bakterie glebowe
Aquificae Reysenbach 2002 – termofilne chemolitotrofy
Bacteroidetes – różnorodna grupa bakterii pasożytniczych, symbiotycznych i wolnożyjących
Chlamydiae Jones 1945 – chlamydie, bezwzględne pasożyty komórek eukariotycznych
Chlorobi Cavalier-Smith 2002 – zielone bakterie siarkowe
Chloroflexi Garrity and Holt 2001 – zielone bakterie bezsiarkowe
Chrysiogenetes Garrity and Holt 2001 – chemolitotrofy
Cyanobacteria (Stanier 1974) Cavalier-Smith 2002 – sinice, bakterie fotosyntetyzujące
Deferribacteres Garrity and Holt 2001 – beztlenowe bakterie wodne
Deinococcus-Thermus Schwartz, 1998 – ekstremofile
Dictyoglomi Saiki et al. 1985 – termofilne chemoorganotrofy
Fibrobacteres Montgomery et al. 1988 – celulolityczne, beztlenowe bakterie żołądkowe
Firmicutes Gibbons & Murray, 1978 – różnorodna grupa pospolitych bakterii
Fusobacteria – beztlenowe heterotrofy, często pasożytnicze
Gemmatimonadetes Zhang i inni, 2003 – Gram-ujemne, tlenowe, pałeczki
Lentisphaerae Cho i inni, 2004
Nitrospira syn. Nitrospirae – Gram-ujemne bakterie utleniające związki azotu
Planctomycetes Schlesner i Stackebrandt 1987, syn. Planctobacteria Cavalier-Smith 2002 – planktobakterie, pozbawione ściany komórkowej, pączkujące
Proteobacteria (Stackebrandt i inni., 1986) Garrity i inni, 2005 – proteobakterie, duża grupa Gram-ujemnych mikroorganizmów
Spirochaetes Cavalier-Smith, 2002 – krętki, syn. Spirochaetae, chemoheterotrofy o spiralnej formie
Thermodesulfobacteria Garrity i Holt, 2001 – termofilne bakterie redukujące siarczany
Thermomicrobia Garrity i Holt, 2001 – termofilne chemoheterotrofy
Thermotogae Reysenbach, 2001 – termofilne, względnie beztlenowe
Verrucomicrobia Hedlund et al. 1998 – bakterie zamieszkujące środowisko wodne, glebowe i układu pokarmowego.
Współżycie z innymi organizmami |

Brodawki korzeniowe u Lotus pedunculatus
Bakterie niejednokrotnie żyją z innymi organizmami. Ich stosunkowo prosta budowa mimo wszystko pozwala im stworzyć bardziej skomplikowanej struktury, z innymi bakteriami (biofilm), lecz znacznie korzystniejsze jest jej utworzenie razem z innymi organizmami. Te różnorodne sposoby współżycia mogą być antagonistyczne i nieantagonistyczne. Wśród najczęściej występujących stosunków międzygatunkowych są symbioza (mutualizm), pasożytnictwo i komensalizm. Bakterie są bardzo małe, co pozwala im na występowanie w dużej ilości w biotopie. Dotyczy to zwłaszcza komensali, które żyją na wszystkich roślinach i zwierzętach, łącznie z ludźmi, u których powodują rozkład niektórych składników potu, przez co ma on charakterystyczny zapach, podobnie jak reszta ciała ludzkiego.
Symbioza |
Pewne bakterie nie są w stanie przetrwać w izolacji, podobnie jak organizmy w których żyją (np. człowiek). Ta interakcja międzygatunkowa charakteryzuje się czerpaniem obustronnych korzyści przez organizmy. Jednym z szczególnych powiązań mutualitycznych jest współżycie bakterii z Archea. Beztlenowe bakterie potrzebują kwasów organicznych, takich jak kwas masłowy czy kwas propionowy. Produkują natomiast wodór, który jest zużywany przez archeowce metanogenowe[128]. Co więcej, gdyby bakterie te nie współżyły z Archea, byłoby to niekorzystne nie tylko dla archeanów (które nie miałyby zapewnionej podaży wodoru), ale również dla samych bakterii, gdyż wysokie stężenie tego pierwiastka jest dla nich toksyczne. Tak więc, gdyby nie było organizmów zużywających wodór, bakterie te nie mogłyby się prawidłowo rozwijać i rozmnażać.
Niektóre drobnoustroje żyjące w ryzosferze przeprowadzają wiązanie azotu cząsteczkowego, przekształcając azot z postaci gazowej w związki chemiczne złożone także z innych pierwiastków[129]. Jest to istotny proces dla roślin oraz organizmów z wyższych poziomów troficznych, gdyż jest to podstawowy sposób przekształcania azotu atmosferycznego w postacie zdatne do asymilacji biologicznej. Współżycie bakterii i roślin określa się bakterioryzą. Wiele innych bakterii znaleziono u zwierząt, np. w ludzkim organizmie jest ich około 1000 gatunków, zwłaszcza w jelitach, gdzie tworzą florę bakteryjną. Pełnią one wiele ważnych funkcji, bez których człowiek nie mógłby żyć. Produkują (syntezują) wiele witamin, w tym witaminę K, kwas foliowy, biotynę. Oprócz tego przetwarzają białka znajdujące się w mleku w kwas mlekowy (głównie Lactobacillus) oraz rozkładają złożone węglowodany do prostszych związków chemicznych[130][131][132]. Obecność tej flory utrudnia rozwój patogenów (głównie na skutek konkurencji, zgodnie z zasadą Gausego) i z tego powodu są one jednymi z najważniejszych probiotyków zalecanych jako suplement diety[133].
Patogenność |
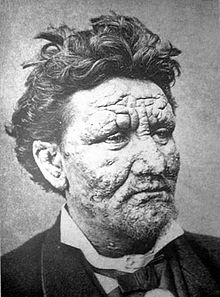
Zdjęcie człowieka w wieku 24 lat chorego na trąd
Jeżeli bakterie szkodzą innym organizmom, wywołując u nich chorobę, określa się je mianem patogenów. Infekcje bakteryjne (bakteriozy) są jedną z przyczyn zgonów ludzi. Wywołują one, między innymi, kiłę, rzeżączkę, dur brzuszny, trąd, cholerę, dżumę, gruźlicę oraz odpowiadają za niektóre zatrucia pokarmowe. Mimo to nie każda bakteria może od razu wywołać infekcję. Wiele bakterii, na przykład Helicobacter pylori, występuje u wielu ludzi, ale tylko czasami ta obecność skutkuje chorobą. Mimo to nosiciele tej bakterii muszą uważać, gdyż zwiększa ona szanse na wystąpienie innych problemów zdrowotnych (np. choroby wrzodowej). Poza tym bakterie mogą także atakować rośliny, co jest powodem wielkich strat w rolnictwie. Mogą one wywołać raka bakteryjnego, zarazę ogniową, kanciastą plamistość, guzowatość korzeni. Atakują też zwierzęta, przez co dodatkowo zagrażają rolnictwu, gdyż mogą wywołać choroby zwierząt gospodarskich (chorobę Johna, mastitis, salmonellozę).
Każdy gatunek bakterii ma charakterystyczne „spektrum” działania na gospodarza. Gronkowiec lub paciorkowiec może np. spowodować infekcję skóry, zapalenie płuc, zapalenie opon mózgowych, a nawet spowodować sepsę, czyli odpowiedź układu immunologicznego (odpornościowego), o bardzo gwałtownym przebiegu, którego skutkiem jest wstrząs i wazodylatacja (zwiotczenie mięśni wokół naczyń krwionośnych), co prowadzi do śmierci organizmu[134]. Dodatkowo wiele bakterii mogących powodować chorobę ustroju przebywa w jelicie, składając się na jego florę, ale nie wywołując infekcji. Istnieją też mikroorganizmy, które niezmiernie rzadko powodują chorobę lub takie, które rzadko pojawiają się w organizmie, jak riketsje, które są pasożytami wewnętrznymi. Jedną z chorób wywoływanych przez riketsje (riketsjoz), czasem uznawaną za najgroźniejszą z tej grupy, jest tyfus plamisty. Inne gatunki mogą wywoływać tyfus plamisty Gór Skalistych, gorączkę okopową i kilka innych chorób. Wśród chlamydii, gromady bakterii będących pasożytami wewnętrznymi, znajduje się gatunek wywołujący u ludzi jeden z typów zapalenia płuc (Chlamydophila pneumoniae) albo zapalenie układu moczowego. Mogą one w skrajnych przypadkach wywoływać nawet chorobę wieńcową serca[135]. W końcu gatunki takie jak Pseudomonas aeruginosa, Burkholderia cenocepacia i Mycobacterium avium są jedną z przyczyn zakażenia oportunistycznego, wywoływanego przez potencjalnie niegroźne bakterie, mogące jednak zbytnio namnożyć się przy osłabionej odporności, czyli u ludzi dotkniętych immunosupresją lub mukowiscydozą[136][137].

Infekcja forsycji spowodowana Agrobacterium tumefaciens
W przypadku infekcji spowodowanej przez bakterie bardzo często do ich zwalczania stosuje się antybiotyki, które mają działanie bakteriobójcze, jeżeli zabijają bakterie lub bakteriostatyczne, jeżeli ich działanie polega na uniemożliwianiu ich prawidłowego wzrostu i rozmnażania. Istnieje bardzo wiele rodzajów antybiotyków, ale każdy jest inhibitorem dla jakiegoś procesu przeprowadzanego tylko przez komórki bakteryjne. Na przykład istnieją antybiotyki o silnej toksyczności, takie jak chloramfenikol lub puromycyna, które działają jednak tylko na rybosomy bakterii, a nie na komórki eukariontów[138]. Antybiotyki są jednak stosowane nie tylko w przypadku ludzi, ale także przy chorobach i profilaktyce zwierząt gospodarczych, co niejednokrotnie może im zaszkodzić, a dodatkowo zwiększa szansę na uodpornienie się szczepu[139]. Rozwojowi bakterii można zapobiegać przez zabiegi antyseptyczne, w tym sterylizację miejsca planowanego nakłucia przed zrobieniem zastrzyku, oraz staranne oczyszczenie samej igły. Również przyrządy stomatologiczne są odpowiednio czyszczone przed wykonaniem zabiegu, by uniknąć możliwości przeniesienia na ich powierzchni patogenów na innych ludzi. Środki do dezynfekcji, na przykład alkohol etylowy, stosowane są, by usunąć wszelkie bakterie z jakiejś powierzchni i zarazem zmniejszyć ryzyko infekcji.
Znaczenie bakterii w przemyśle |

Ogórki kiszone uzyskiwane dzięki działaniu bakterii z rodzaju Lactobacillus
Bakterie, między innymi Lactobacillus znajdujące się w mleku po dodaniu do drożdży, są stosowane od tysięcy lat przy wytwarzaniu różnych produktów spożywczych (kiszonej kapusty, sera, sosu sojowego, wina, octu, jogurtu)[95][140][141].
Zdolność bakterii do degradacji (rozkładania) wielu związków organicznych jest niezwykle przydatna dla człowieka i z tego powodu są one wykorzystywane w przemyśle i gospodarce. Mikroorganizmy zdolne do rozkładania węglowodorów są używane do likwidacji wycieków ropy naftowej lub innych olejów z tankowców, dzięki czemu ułatwiają usuwanie skutków wielu katastrof ekologicznych[142]. Przykładem zastosowania bioremediacji była likwidacja wylewu ropy do morza przy Zatoce Księcia Williama po katastrofie tankowca Exxon Valdez w 1989 roku. Bakterie zostały użyte by zwiększyć skuteczność usuwania skutków katastrofy ekologicznej. Dodatkowo rozrzucono je także na plażach, gdzie były w stanie zutylizować ropę, która wsiąkła w piasek. Stosuje się je także do unieszkodliwiania toksycznych odpadów przemysłowych[143]. W przemyśle chemicznym bakterie są bardzo ważne przy produkcji enancjomerów oraz wytwarzaniu czystych substancji chemicznych mających służyć jako środki do wytwarzania leków lub agrochemikaliów[144].
Bakterie mogą również zostać użyte do biologicznego zwalczania szkodników w miejsce pestycydów. Zwykle używa się Bacillus thuringiensis (zwaną również BT), Gram-dodatnią bakterię, która występuje w glebach. Jej podgatunki są używane do zwalczania gąsienic motyli. Są one składnikiem środków takich jak Dipel i Thuricide[145]. Z powodu nietoksycznych składników oraz zastosowania bakterii groźnych tylko dla niektórych organizmów są one bezpieczne dla środowiska. Bardzo rzadko dochodzi do sytuacji, w której środki te mogą zaszkodzić ludziom lub zwierzętom pożytecznym, oraz samym roślinom[146][147].
Z powodu zdolności bakterii do szybkiego wzrostu są one cennym obiektem badań molekularnych. Przez modyfikowanie ich struktury genetycznej i sprawdzeniu jak zmiany te kształtują fenotyp populacji, naukowcy mogą sprawdzić jakie geny, enzymy i funkcje metabolityczne charakteryzują poszczególne bakterie oraz jak je zmieniać. Wiedzę tę można użyć przy modyfikowaniu bardziej złożonych organizmów, np. roślin lub zwierząt[148]. Dzięki temu można zrozumieć dokładnie jak bardzo złożony jest świat żywych organizmów i jak proste pierwiastki były w stanie stworzyć życie[149][150]. Poznanie tych funkcji umożliwia także bioinżynierii produkcję insuliny, witamin, przeciwciał oraz innych substancji[151][152]. Są one wytwarzane, po modyfikacji genetycznej bakterii, która na podstawie nowych genów produkuje np. hormony.
Przypisy |
↑ HN. Schulz, BB. Jorgensen. Big bacteria.. „Annu Rev Microbiol”. 55, s. 105-37, 2001. DOI: 10.1146/annurev.micro.55.1.105. PMID: 11544351.
↑ JK. Fredrickson, JM. Zachara, DL. Balkwill, D. Kennedy i inni. Geomicrobiology of High-Level Nuclear Waste-Contaminated Vadose Sediments at the Hanford Site, Washington State. „Applied and Environmental Microbiology”. 70 (7), s. 4230–41, lipiec 2004. DOI: 10.1128/AEM.70.7.4230-4241.2004. PMID: 15240306. [dostęp 2008-08-30].
↑ Whitman WB., Coleman DC., Wiebe WJ. Prokaryotes: the unseen majority. „Proceedings of the National Academy of Sciences of the United States of America”. 95 (12), s. 6578–83, czerwiec 1998. PMID: 9618454.
↑ ab Rappé MS., Giovannoni SJ. The uncultured microbial majority. „Annual Review of Microbiology”. 57, s. 369–94, 2003. DOI: 10.1146/annurev.micro.57.030502.090759. PMID: 14527284.
↑ Porter JR., van Leeuwenhoek A. Antony van Leeuwenhoek: tercentenary of his discovery of bacteria. „Bacteriological Reviews”. 40 (2), s. 260–9, czerwiec 1976. PMID: 786250.
↑ Anthony Leewenhoeck. An Abstract of a Letter from Mr. Anthony Leewenhoeck at Delft, Dated Sep. 17. 1683. Containing Some Microscopical Observations, about Animals in the Scurf of the Teeth, the Substance Call’d Worms in the Nose, the Cuticula Consisting of Scales. „Philosophical Transactions”. 14, s. 568-574, 1684. Royal Society Publishing. DOI: 10.1098/rstl.1684.0030. [dostęp 2008-11-22].
↑ Online Etymology Dictionary. [dostęp 2008-09-03].
↑ Louis Pasteur, Jourbert Chamberland. The Germ Theory and Its Applications to Medicine and Surgery. „Comptes Rendus de l' Academie des Sciences”. lxxxvi, s. 1037-43, 1878-04-29. [dostęp 2008-09-03].
↑ The Nobel Prize in Physiology or Medicine 1905. [dostęp 2008-09-03].
↑ O’Brien SJ., Goedert JJ. HIV causes AIDS: Koch’s postulates fulfilled. „Current Opinion in Immunology”. 8 (5), s. 613–8, październik 1996. PMID: 8902385.
↑ Thurston AJ. Of blood, inflammation and gunshot wounds: the history of the control of sepsis. „The Australian and New Zealand Journal of Surgery”. 70 (12), s. 855–861, 2000. DOI: 10.1046/j.1440-1622.2000.01983.x. PMID: 11167573.
↑ Schwartz RS., Ehrlich P. Paul Ehrlich’s magic bullets. „The New England Journal of Medicine”. 350 (11), s. 1079–1080, 2004. DOI: 10.1056/NEJMp048021. PMID: 15014180.
↑ Amanda Yarnell. Salvarsan. „Chemical & Engineering News”. 83 (25), 20 czerwca 2005.
↑ Paul Ehrlich: The Nobel Prize in Physiology or Medicine 1908 (ang.). Nobelprize.org. [dostęp 2008-09-05].
↑ Woese CR., Fox GE. Phylogenetic structure of the prokaryotic domain: the primary kingdoms. „Proceedings of the National Academy of Sciences of the United States of America”. 74 (11), s. 5088–5090, 1977. DOI: 10.1073/pnas.74.11.5088. PMID: 270744. PMCID: PMC432104.
↑ ab Woese CR., Kandler O., Wheelis ML. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya. „Proceedings of the National Academy of Sciences of the United States of America”. 87 (12), s. 4576–4579, 1990. DOI: 10.1073/pnas.87.12.4576. PMID: 2112744. PMCID: PMC54159.
↑ Heide N. Schulz, Bo Barker Jørgensen. Big Bacteria. „Annual Review of Microbiology”. 55, s. 105-137, 2001. DOI: 10.1146/annurev.micro.55.1.105.
↑ Robertson J, Gomersall M, Gill P. Mycoplasma hominis: growth, reproduction, and isolation of small viable cells. „J Bacteriol.”. 2 (124), s. 1007–18, 1975. PMID: 1102522. PMCID: PMC235991.
↑ Velimirov, B. Nanobacteria, Ultramicrobacteria and Starvation Forms: A Search for the Smallest Metabolizing Bacterium. „Microbes and Environments”. 2 (16), s. 67–77, 2001. DOI: 10.1264/jsme2.2001.67.
↑ Ingo Fritz, Carsten Strömpl, Wolf-Rainer Abraham. Phylogenetic relationships of the genera Stella, Labrys and Angulomicrobium within the ‘Alphaproteobacteria’ and description of Angulomicrobium amanitiforme sp. nov.. „International Journal of Systematic and Evolutionary Microbiology”. 54, s. 651-657, 2004. DOI: 10.1099/ijs.0.02746-0. PMID: 15143003.
↑ Kevin D. Young. The Selective Value of Bacterial Shape. „Microbiology and Molecular Biology Reviews”. 70 (3), s. 660-703, sierpień 2006. DOI: 10.1128/MMBR.00001-06. [dostęp 2008-11-22].
↑ ab Douwes KE, Schmalzbauer E, Linde HJ, Reisberger EM i inni. Branched filaments no fungus, ovoid bodies no bacteria: Two unusual cases of mycetoma. „Journal of the American Academy of Dermatology”. 49 (2), s. 170-173, 2003. PMID: 12894113.
↑ Donlan RM. Biofilms: microbial life on surfaces. „Emerging Infectious diseases”. 8 (9), s. 881-90, 2002. DOI: 10.3201/eid0809.020063. PMID: 12194761. PMCID: PMC2732559.
↑ Rodney M. Donlan, J. William Costerton. Biofilms: Survival Mechanisms of Clinically Relevant Microorganisms. „Clinical Microbiology Reviews”. 15 (2), s. 167-193, Apr 2002. DOI: 10.1128/CMR.15.2.167-193.2002. PMID: 11932229. PMCID: PMC118068.
↑ ab Lawrence J. Shimkets. Intercellular Signaling During Frutining-Body Development of Myxococcus xanthus. „Annual Review of Microbiology”. 53, s. 525-549, 1999. DOI: 10.1146/annurev.micro.53.1.525.
↑ ab Witold Mizerski: Tablice biologiczne, praca zbiorowa. Warszawa: Adamantan, 2013, s. 55. ISBN 978-83-7350-243-7.
↑ ab Dale Kaiser. Signaling in Myxobacteria. „Annual Review of Microbiology”. 58, s. 75-98, 2004. DOI: 10.1146/annurev.micro.58.030603.123620. PMID: 15487930.
↑ JM Berg, JL Tymoczko, L Stryer: Biochemistry. New York: W.H. Freeman and Company, 2002. ISBN 0-7167-4955-6.
↑ Yu-Ling Shih, Lawrence Rothfield. The Bacterial Cytoskeleton. „Microbiology and Molecular Biology Review”. 70 (3), s. 729–754, 2006. DOI: 10.1128/MMBR.00017-06. PMID: 16959967. PMCID: PMC1594594.
↑ Z. Gitai. The New Bacterial Cell Biology: Moving Parts and Subcellular Architecture. „Cell”. 120 (5), s. 577-586, 2005-03-11. DOI: 10.1016/j.cell.2005.02.026.
↑
Kerfeld CA, Sawaya MR, Tanaka S, et al. Protein structures forming the shell of primitive bacterial organelles. „Science (journal)”. 5736 (309), s. 936–8, sierpień 2005. DOI: 10.1126/science.1113397. PMID: 16081736.
↑ Yeates TO, Kerfeld CA, Heinhorst S, Cannon GC, Shively JM. Protein-based organelles in bacteria: carboxysomes and related microcompartments. „Nat. Rev. Microbiol.”, s. 681–691, sierpień 2008. DOI: 10.1038/nrmicro1913. PMID: 18679172.
↑ Bobik, T. A. Polyhedral organelles compartmenting bacterial metabolic processes. „Applied Microbiology and Biotechnology”. 5 (70), s. 517–525, 2006. DOI: 10.1007/s00253-005-0295-0.
↑
Bryant DA, Frigaard NU. Prokaryotic photosynthesis and phototrophy illuminated. „Trends Microbiol.”. 11 (14), s. 488, 2006. DOI: 10.1016/j.tim.2006.09.001.
↑ Thanbichler M, Wang S, Shapiro L. The bacterial nucleoid: a highly organized and dynamic structure. „J Cell Biochem”. 3 (96), s. 506–21, 2005. DOI: 10.1002/jcb.20519. PMID: 15988757.
↑ Fuerst J. Intracellular compartmentation in planctomycetes. „Annu Rev Microbiol”, s. 299–328, 2005. DOI: 10.1146/annurev.micro.59.030804.121258. PMID: 15910279.
↑ ab Walsby A. Gas vesicles. „Microbiol Rev”. 1 (58), s. 94–144, 1994. PMID: 8177173.
↑
Yeo M, Chater K. The interplay of glycogen metabolism and differentiation provides an insight into the developmental biology of Streptomyces coelicolor. „Microbiology”. Pt 3 (151), s. 855–61, 2005. DOI: 10.1099/mic.0.27428-0. PMID: 15758231.
↑ Shiba T, Tsutsumi K, Ishige K, Noguchi T. Inorganic polyphosphate and polyphosphate kinase: their novel biological functions and applications. „Biochemistry (Mosc)”. 3 (65), s. 315–23, 2000. PMID: 10739474.
↑ Brune DC.. Isolation and characterization of sulfur globule proteins from Chromatium vinosum and Thiocapsa roseopersicina. „Arch Microbiol”. 163 (6), s. 391–99, 1995. DOI: 10.1007/BF00272127. PMID: 7575095.
↑
D. Kadouri, E. Jurkevitch, Y. Okon, S. Castro-Sowinski. Ecological and agricultural significance of bacterial polyhydroxyalkanoates. „Crit Rev Microbiol”. 31 (2), s. 55–67, 2005. DOI: 10.1080/10408410590899228. PMID: 15986831.
↑ Antoni Różalski. Ćwiczenia z mikrobiologii ogólnej. Skrypt dla studentów biologii.. , s. 117, 1996.
↑ van Heijenoort J. Formation of the glycan chains in the synthesis of bacterial peptidoglycan. „Glycobiology”. 3 (11), s. 25R-36R, 2001. DOI: 10.1093/glycob/11.3.25R. PMID: 11320055.
↑ Koch A. Bacterial wall as target for attack: past, present, and future research. „Clin Microbiol Rev”. 4 (16), s. 673–87, 2003. DOI: 10.1128/CMR.16.4.673-687.2003. PMID: 14557293.
↑ Hans Christian Gram: Über die isolierte Färbung der Schizomyceten in Schnitt- und Trockenpräparaten. 1884.
↑
Hugenholtz P. Exploring prokaryotic diversity in the genomic era. „Genome Biol”. 2 (3), s. REVIEWS0003, 2002. DOI: 10.1186/gb-2002-3-2-reviews0003. PMID: 11864374.
↑ Walsh F, Amyes S. Microbiology and drug resistance mechanisms of fully resistant pathogens. „Curr Opin Microbiol”. 5 (7), s. 439–44, 2004. DOI: 10.1016/j.mib.2004.08.007. PMID: 15451497.
↑ Engelhardt H, Peters J. Structural research on surface layers: a focus on stability, surface layer homology domains, and surface layer-cell wall interactions. „J Struct Biol”. 2–3 (124), s. 276–302, 1998. DOI: 10.1006/jsbi.1998.4070. PMID: 10049812.
↑
Beveridge T, Pouwels P, Sára M, Kotiranta A, Lounatmaa K, Kari K, Kerosuo E, Haapasalo M, Egelseer E, Schocher I, Sleytr U, Morelli L, Callegari M, Nomellini J, Bingle W, Smit J, Leibovitz E, Lemaire M, Miras I, Salamitou S, Béguin P, Ohayon H, Gounon P, Matuschek M, Koval S. Functions of S-layers. „FEMS Microbiol Rev”. 1–2 (20), s. 99–149, 1997. PMID: 9276929.
↑ Fizjologia z elementami anatomii i histologii. W: Jerzy Błoszyk, Małgorzata Maćkowiak, Anna (biologia) Michalak: Biologia: jedność i różnorodność. Warszawa: Wydawnictwo Szkolne PWN, 2008, s. 474. ISBN 978-83-7446-134-4.
↑ Beachey E. Bacterial adherence: adhesin-receptor interactions mediating the attachment of bacteria to mucosal surface. „J Infect Dis”. 3 (143), s. 325–45, 1981. PMID: 7014727.
↑
Silverman P. Towards a structural biology of bacterial conjugation. „Mol Microbiol”. 3 (23), s. 423–9, 1997. DOI: 10.1046/j.1365-2958.1997.2411604.x. PMID: 9044277.
↑
Stokes R, Norris-Jones R, Brooks D, Beveridge T, Doxsee D, Thorson L. The glycan-rich outer layer of the cell wall of Mycobacterium tuberculosis acts as an antiphagocytic capsule limiting the association of the bacterium with macrophages. „Infect Immun”. 10 (72), s. 5676–86, 2004. DOI: 10.1128/IAI.72.10.5676-5686.2004. PMID: 15385466.
↑ Daffé M, Etienne G. The capsule of Mycobacterium tuberculosis and its implications for pathogenicity. „Tuber Lung Dis”. 3 (79), s. 153–69, 1999. DOI: 10.1054/tuld.1998.0200. PMID: 10656114.
↑ Finlay B, Falkow S. Common themes in microbial pathogenicity revisited. „Microbiol Mol Biol Rev”. 2 (61), s. 136–69, 1997. PMID: 9184008.
↑
Nicholson W, Munakata N, Horneck G, Melosh H, Setlow P. Resistance of Bacillus endospores to extreme terrestrial and extraterrestrial environments. „Microbiol Mol Biol Rev”. 3 (64), s. 548–572, 2000. DOI: 10.1128/MMBR.64.3.548-572.2000. PMID: 10974126. PMCID: PMC99004.
↑ Siunov A, Nikitin D, Suzina N, Dmitriev V, Kuzmin N, Duda V. Phylogenetic status of Anaerobacter polyendosporus, an anaerobic, polysporogenic bacterium. „Int J Syst Bacteriol”. s. 1119–24. PMID: 10425769.
↑ Nicholson W, Fajardo-Cavazos P, Rebeil R, Slieman T, Riesenman P, Law J, Xue Y. Bacterial endospores and their significance in stress resistance. „Antonie Van Leeuwenhoek”. 1–4 (81), s. 27–32, 2002. DOI: 10.1023/A:1020561122764. PMID: 12448702.
↑ Vreeland R, Rosenzweig W, Powers D. Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal. „Nature”. 6806 (407), s. 897–900, 2000. DOI: 10.1038/35038060. PMID: 11057666.
↑ Cano R, Borucki M. Revival and identification of bacterial spores in 25- to 40-million-year-old Dominican amber. „Science”. 5213 (268), s. 1060–4, 1995. DOI: 10.1126/science.7538699. PMID: 7538699.
↑ Nicholson W, Schuerger A, Setlow P. The solar UV environment and bacterial spore UV resistance: considerations for Earth-to-Mars transport by natural processes and human spaceflight. „Mutat Res”. 1–2 (571), s. 249–64, 2005. PMID: 15748651.
↑ Hatheway C. Toxigenic clostridia. „Clin Microbiol Rev”. 1 (3), s. 66–98, 1990. PMID: 2404569.
↑ Nealson K. Post-Viking microbiology: new approaches, new data, new insights. „Orig Life Evol Biosph”. 1 (29), s. 73–93, 1999. DOI: 10.1023/A:1006515817767. PMID: 11536899.
↑ Xu J. Microbial ecology in the age of genomics and metagenomics: concepts, tools, and recent advances. „Mol Ecol”. 7 (15), s. 1713–31, 2006. DOI: 10.1111/j.1365-294X.2006.02882.x. PMID: 16689892.
↑ Zillig W. Comparative biochemistry of Archaea and Bacteria. „Curr Opin Genet Dev”. 4 (1), s. 544–51, 1991. DOI: 10.1016/S0959-437X(05)80206-0. PMID: 1822288.
↑ Hellingwerf K, Crielaard W, Hoff W, Matthijs H, Mur L, van Rotterdam B. Photobiology of bacteria. „Antonie Van Leeuwenhoek”. 4 (65), s. 331–47, 1994. DOI: 10.1007/BF00872217. PMID: 7832590.
↑ Zumft W. Cell biology and molecular basis of denitrification. „Microbiol Mol Biol Rev”. 4 (61), s. 533–616, 1997. PMID: 9409151. PMCID: PMC232623.
↑ Drake H, Daniel S, Küsel K, Matthies C, Kuhner C, Braus-Stromeyer S. Acetogenic bacteria: what are the in situ consequences of their diverse metabolic versatilities?. „Biofactors”. 1 (6), s. 13–24, 1997. DOI: 10.1002/biof.5520060103. PMID: 9233536.
↑ ab FMM Morel, Kraepiel AML, Amyot M. The chemical cycle and bioaccumulation of mercury. „Annual Review of Ecological Systems”, s. 543–566, 1998. DOI: 10.1146/annurev.ecolsys.29.1.543.
↑ Zehr J, Jenkins B, Short S, Steward G. Nitrogenase gene diversity and microbial community structure: a cross-system comparison. „Environ Microbiol”. 7 (5), s. 539–554, 2003. DOI: 10.1046/j.1462-2920.2003.00451.x. PMID: 12823187.
↑ Koch A. Control of the bacterial cell cycle by cytoplasmic growth. „Crit Rev Microbiol”. 1 (28), s. 61–77, 2002. DOI: 10.1080/1040-840291046696. PMID: 12003041.
↑ Eagon R. Pseudomonas natriegens, a marine bacterium with a generation time of less than 10 minutes. „J Bacteriol”. s. 736–737. PMID: 13888946. PMCID: PMC279347.
↑ abc Thomson R, Bertram H. Laboratory diagnosis of central nervous system infections. „Infect Dis Clin North Am”. 4 (15), s. 1047–1071, 2001. DOI: 10.1016/S0891-5520(05)70186-0. PMID: 11780267.
↑ Paerl H, Fulton R, Moisander P, Dyble J. Harmful freshwater algal blooms, with an emphasis on cyanobacteria. „ScientificWorldJournal”, s. 76–113, 2001. DOI: 10.1100/tsw.2001.16. PMID: 12805693.
↑ Prats C., López D., Giró A., Ferrer J., Valls J. Individual-based modelling of bacterial cultures to study the microscopic causes of the lag phase. „Journal of theoretical biology”. 4 (241), s. 939–53, sierpień 2006. DOI: 10.1016/j.jtbi.2006.01.029. PMID: 16524598.
↑ Hecker M, Völker U. General stress response of Bacillus subtilis and other bacteria. „Adv Microb Physiol”, s. 35–91, 2001. DOI: 10.1016/S0065-2911(01)44011-2. PMID: 11407115.
↑ Stewart EJ., Madden R., Paul G., Taddei F. Aging and death in an organism that reproduces by morphologically symmetric division.. „PLoS biology”. 2 (3), s. e45, luty 2005. DOI: 10.1371/journal.pbio.0030045. PMID: 15685293.
↑
Challis G, Hopwood D. Synergy and contingency as driving forces for the evolution of multiple secondary metabolite production by Streptomyces species. „Proc Natl Acad Sci U S a”, s. 14555–61, 2003. DOI: 10.1073/pnas.1934677100. PMID: 12970466.
↑ Kooijman S, Auger P, Poggiale J, Kooi B. Quantitative steps in symbiogenesis and the evolution of homeostasis. „Biol Rev Camb Philos Soc”. 3 (78), s. 435–63, 2003. DOI: 10.1017/S1464793102006127. PMID: 14558592.
↑ Prats C, López D, Giró A, Ferrer J, Valls J. Individual-based modelling of bacterial cultures to study the microscopic causes of the lag phase. „J Theor Biol”. 4 (241), s. 939–53, 2006. PMID: 16524598.
↑ Nakabachi A, Yamashita A, Toh H, Ishikawa H, Dunbar H, Moran N, Hattori M. The 160-kilobase genome of the bacterial endosymbiont Carsonella. „Science”. 5797 (314), s. 267, 2006. DOI: 10.1126/science.1134196. PMID: 17038615.
↑ Schneiker, S., O. Perlova, et al.. Complete genome sequence of the myxobacterium Sorangium cellulosum.. „Nature Biotechnology”. 25 (11), s. 1281-89, 2007. DOI: 10.1038/nbt1354.
↑ Hinnebusch J, Tilly K. Linear plasmids and chromosomes in bacteria. „Mol Microbiol”. 5 (10), s. 917–22, 1993. DOI: 10.1111/j.1365-2958.1993.tb00963.x. PMID: 7934868.
↑ Belfort M, Reaban ME, Coetzee T, Dalgaard JZ. Prokaryotic introns and inteins: a panoply of form and function. „J. Bacteriol.”. 14 (177), s. 3897–903, 1995. PMID: 7608058.
↑ Brüssow H, Canchaya C, Hardt W. Phages and the evolution of bacterial pathogens: from genomic rearrangements to lysogenic conversion. „Microbiol Mol Biol Rev”. 3 (68), s. 560–602, 2004. DOI: 10.1128/MMBR.68.3.560-602.2004. PMID: 15353570.
↑ Bickle TA, Krüger DH. Biology of DNA restriction. „Microbiol. Rev.”. 2 (57), s. 434–50, czerwiec 1993. PMID: 8336674.
↑ Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes. „Science (journal)”. 5819 (315), s. 1709–12, marzec 2007. DOI: 10.1126/science.1138140. PMID: 17379808.
↑ Brouns SJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. „Science (journal)”. 5891 (321), s. 960–4, sierpień 2008. DOI: 10.1126/science.1159689. PMID: 18703739.
↑ Denamur E, Matic I. Evolution of mutation rates in bacteria. „Mol Microbiol”. 4 (60), s. 820–7, 2006. DOI: 10.1111/j.1365-2958.2006.05150.x. PMID: 16677295.
↑ Wright B. Stress-directed adaptive mutations and evolution. „Mol Microbiol”. 3 (52), s. 643–50, 2004. DOI: 10.1111/j.1365-2958.2004.04012.x. PMID: 15101972.
↑ Davison J. Genetic exchange between bacteria in the environment. „Plasmid”. 2 (42), s. 73–91, 1999. DOI: 10.1006/plas.1999.1421. PMID: 10489325.
↑ Hastings P, Rosenberg S, Slack A. Antibiotic-induced lateral transfer of antibiotic resistance. „Trends Microbiol”. 9 (12), s. 401–4, 2004. DOI: 10.1016/j.tim.2004.07.003. PMID: 15337159.
↑ abc Bardy SL., Ng SY., Jarrell KF. Prokaryotic motility structures. „Microbiology (Reading, England)”. Pt 2 (149), s. 295–304, luty 2003. PMID: 12624192.
↑ Merz A, So M, Sheetz M. Pilus retraction powers bacterial twitching motility. „Nature”. 6800 (407), s. 98–102, 2000. DOI: 10.1038/35024105. PMID: 10993081.
↑ ab Beata Bednarczuk: Słownik bakterii. Warszawa: adamantan, 2008, s. 247. ISBN 978-83-7350-076-1.
↑ Wu M, Roberts J, Kim S, Koch D, DeLisa M. Collective bacterial dynamics revealed using a three-dimensional population-scale defocused particle tracking technique. „Appl Environ Microbiol”. 7 (72), s. 4987–4994, 2006. DOI: 10.1128/AEM.00158-06. PMID: 16820497.
↑ Goldberg MB. Actin-based motility of intracellular microbial pathogens. „Microbiol Mol Biol Rev”. 4 (65), s. 595–626, 2001. DOI: 10.1128/MMBR.65.4.595-626.2001. PMID: 11729265.
↑ Lux R, Shi W. Chemotaxis-guided movements in bacteria. „Crit Rev Oral Biol Med”. 4 (15), s. 207–20, 2004. PMID: 15284186.
↑ Frankel R, Bazylinski D, Johnson M, Taylor B. Magneto-aerotaxis in marine coccoid bacteria. „Biophys J”. 2 (73), s. 994–1000, 1997. DOI: 10.1016/S0006-3495(97)78132-3. PMID: 9251816.
↑
Schopf J. Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic. „Proc Natl Acad Sci U S a”. 15 (91), s. 6735–42, 1994. DOI: 10.1073/pnas.91.15.6735. PMID: 8041691.
↑
DeLong E, Pace N. Environmental diversity of bacteria and archaea. „Syst Biol”. 4 (50), s. 470–78, 2001. DOI: 10.1080/106351501750435040. PMID: 12116647.
↑
Brown JR, Doolittle WF. Archaea and the prokaryote-to-eukaryote transition. „Microbiol. Mol. Biol. Rev.”. 4 (61), s. 456–502, 1997. PMID: 9409149.
↑ Di Giulio M. The universal ancestor and the ancestor of bacteria were hyperthermophiles. „J Mol Evol”. 6 (57), s. 721–30, 2003. DOI: 10.1007/s00239-003-2522-6. PMID: 14745541.
↑
Battistuzzi F, Feijao A, Hedges S. A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land. „BMC Evol Biol”, s. 44, 2004. DOI: 10.1186/1471-2148-4-44. PMID: 15535883.
↑ Dyall S, Brown M, Johnson P. Ancient invasions: from endosymbionts to organelles. „Science”. 5668 (304), s. 253–7, 2004. DOI: 10.1126/science.1094884. PMID: 15073369.
↑
Poole A, Penny D. Evaluating hypotheses for the origin of eukaryotes. „Bioessays”. 1 (29), s. 74–84, 2007. DOI: 10.1002/bies.20516. PMID: 17187354.
↑ Ebersold HR, Cordier JL, Lüthy P. Bacterial mesosomes: method dependent artifacts. „Archives of Microbiology”. 1981. 130. s. 19–22. DOI: 10.1007/BF00527066. PMID: 6796029.
↑
Lang B, Gray M, Burger G. Mitochondrial genome evolution and the origin of eukaryotes. „Annu Rev Genet”, s. 351–97, 1999. DOI: 10.1146/annurev.genet.33.1.351. PMID: 10690412.
↑ Schulz H, Jorgensen B. Big bacteria. „Annu Rev Microbiol”, s. 105–37, 2001. DOI: 10.1146/annurev.micro.55.1.105. PMID: 11544351.
↑ Boucher Y, Douady CJ, Papke RT, Walsh DA, Boudreau ME, Nesbo CL, Case RJ, Doolittle WF. Lateral gene transfer and the origins of prokaryotic groups. „Annu Rev Genet”, s. 283–328, 2003. DOI: 10.1146/annurev.genet.37.050503.084247. PMID: 14616063.
↑ Olsen G, Woese C, Overbeek R. The winds of (evolutionary) change: breathing new life into microbiology. „J Bacteriol”. 176 (1), s. 1–6, 1994. PMID: 8282683.
↑ Gupta R. The natural evolutionary relationships among prokaryotes. „Crit Rev Microbiol”. 2 (26), s. 111–31, 2000. DOI: 10.1080/10408410091154219. PMID: 10890353.
↑ Cavalier-Smith T. The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification. „Int J Syst Evol Microbiol”. Pt 1 (52), s. 7–76, 2002. PMID: 11837318.
↑ Hans Christian Gram: Über die isolierte Färbung der Schizomyceten in Schnitt- und Trockenpräparaten. Fortschr. Med., 1884.
↑ Woods G, Walker D. Detection of infection or infectious agents by use of cytologic and histologic stains. „Clin Microbiol Rev”. 3 (9), s. 382–404, 1996. PMID: 8809467. PMCID: PMC172900.
↑ Weinstein M. Clinical importance of blood cultures. „Clin Lab Med”. 1 (14), s. 9–16, 1994. PMID: 8181237.
↑ Louie M, Louie L, Simor AE. The role of DNA amplification technology in the diagnosis of infectious diseases. „CMAJ”. 3 (163), s. 301–309, 2000. PMID: 10951731.
↑ Oliver J. The viable but nonculturable state in bacteria. „J Microbiol”. 43 Spec No, s. 93–100, Feb 2005. PMID: 15765062.
↑ Numbers of Living Species in Australia and the World 2nd edition. [dostęp kwicień 2007].
↑ Curtis T, Sloan W, Scannell J. Estimating prokaryotic diversity and its limits. „Proc Natl Acad Sci U S a”. 99 (16), s. 10494–9, 2002. DOI: 10.1073/pnas.142680199. PMID: 12097644.
↑ Schloss P, Handelsman J. Status of the microbial census. „Microbiol Mol Biol Rev”. 4 (68), s. 686–91, 2004. DOI: 10.1128/MMBR.68.4.686-691.2004. PMID: 15590780.
↑ Richard Harder: Systematyka. W: Eduard Strasburger, i in.: Botanika: podręcznik dla szkół wyższych. Wyd. 2 pol. według 28 oryg. Warszawa: PWRiL, 1967.
↑ Zbigniew Podbielkowski, Irena Rejment-Grochowska, Alina Skirgiełło: Rośliny zarodnikowe. Wyd. 2. Warszawa: PWN, 1980.
↑ Approved Lists of Bacterial Names (ang.). American Society for Microbiology. [dostęp 2009-03-11].
↑ International Code of Nomenclature of Bacteria (1990 Revision) (ang.). International Union of Microbiological Societies. [dostęp 2009-03-11].
↑ J.P. Euzéby: Classification of domains and phyla – Hierarchical classification of prokaryotes (bacteria).
↑ Bergey’s Manual Trust Bergey’s Manual Trust.
↑ Stams A, de Bok F, Plugge C, van Eekert M, Dolfing J, Schraa G. Exocellular electron transfer in anaerobic microbial communities. „Environ Microbiol”. 3 (8), s. 371–82, 2006. DOI: 10.1111/j.1462-2920.2006.00989.x. PMID: 16478444.
↑ Barea J, Pozo M, Azcón R, Azcón-Aguilar C. Microbial co-operation in the rhizosphere. „J Exp Bot”. 417 (56), s. 1761–78, 2005. DOI: 10.1093/jxb/eri197. PMID: 15911555.
↑ O’Hara A, Shanahan F. The gut flora as a forgotten organ. „EMBO Rep”. 7 (7), s. 688–93, 2006. DOI: 10.1038/sj.embor.7400731. PMID: 16819463.
↑ Zoetendal E, Vaughan E, de Vos W. A microbial world within us. „Mol Microbiol”. 6 (59), s. 1639–50, 2006. DOI: 10.1111/j.1365-2958.2006.05056.x. PMID: 16553872.
↑ Gorbach S. Lactic acid bacteria and human health. „Ann Med”. 1 (22), s. 37–41, 1990. DOI: 10.3109/07853899009147239. PMID: 2109988.
↑ Salminen S, Gueimonde M, Isolauri E. Probiotics that modify disease risk. „J Nutr”. 5 (135), s. 1294–8, 2005. PMID: 15867327.
↑ Fish D. Optimal antimicrobial therapy for sepsis. „Am J Health Syst Pharm”. s. S13–9. PMID: 11885408.
↑ Belland R, Ouellette S, Gieffers J, Byrne G. Chlamydia pneumoniae and atherosclerosis. „Cell Microbiol”. 2 (6), s. 117–27, 2004. DOI: 10.1046/j.1462-5822.2003.00352.x. PMID: 14706098.
↑ Heise E. Diseases associated with immunosuppression. „Environ Health Perspect”, s. 9–19, 1982. DOI: 10.2307/3429162. PMID: 7037390. PMCID: PMC1568899.
↑ L Saiman. Microbiology of early CF lung disease. „Paediatr Respir Rev.”. 5 Suppl A. s. S367–369. PMID: 14980298.
↑ Yonath A, Bashan A. Ribosomal crystallography: initiation, peptide bond formation, and amino acid polymerization are hampered by antibiotics. „Annu Rev Microbiol”, s. 233–51, 2004. DOI: 10.1146/annurev.micro.58.030603.123822. PMID: 15487937.
↑ Khachatourians G. Agricultural use of antibiotics and the evolution and transfer of antibiotic-resistant bacteria. „CMAJ”. 9 (159), s. 1129–1136, 1998. PMID: 9835883. PMCID: PMC1229782.
↑ Johnson M, Lucey J. Major technological advances and trends in cheese. „J Dairy Sci”. 4 (89), s. 1174–8, 2006. PMID: 16537950.
↑ Hagedorn S, Kaphammer B. Microbial biocatalysis in the generation of flavor and fragrance chemicals. „Annu. Rev. Microbiol.”, s. 773–800, 1994. DOI: 10.1146/annurev.mi.48.100194.004013. PMID: 7826026.
↑ Cohen Y. Bioremediation of oil by marine microbial mats. „Int Microbiol”. 4 (5), s. 189–93, 2002. DOI: 10.1007/s10123-002-0089-5. PMID: 12497184.
↑ Neves LC, Miyamura TT, Moraes DA, Penna TC, Converti A. Biofiltration methods for the removal of phenolic residues. „Appl. Biochem. Biotechnol.”, s. 130–52, 2006. DOI: 10.1385/ABAB:129:1:130. PMID: 16915636.
↑ Liese A, Filho M. Production of fine chemicals using biocatalysis. „Curr Opin Biotechnol”. 6 (10), s. 595–603, 1999. DOI: 10.1016/S0958-1669(99)00040-3. PMID: 10600695.
↑ Aronson AI, Shai Y. Why Bacillus thuringiensis insecticidal toxins are so effective: unique features of their mode of action. „FEMS Microbiol. Lett.”. 1 (195), s. 1–8, 2001. DOI: 10.1111/j.1574-6968.2001.tb10489.x. PMID: 11166987.
↑ Bozsik A. Susceptibility of adult Coccinella septempunctata (Coleoptera: Coccinellidae) to insecticides with different modes of action. „Pest Manag Sci”. 7 (62), s. 651–4, 2006. DOI: 10.1002/ps.1221. PMID: 16649191.
↑ Chattopadhyay A, Bhatnagar N, Bhatnagar R. Bacterial insecticidal toxins. „Crit Rev Microbiol”. 1 (30), s. 33–54, 2004. DOI: 10.1080/10408410490270712. PMID: 15116762.
↑ Serres M, Gopal S, Nahum L, Liang P, Gaasterland T, Riley M. A functional update of the Escherichia coli K-12 genome. „Genome Biol”. 9 (2), s. RESEARCH0035, 2001. DOI: 10.1186/gb-2001-2-9-research0035. PMID: 11574054.
↑ Almaas E, Kovács B, Vicsek T, Oltvai Z, Barabási A. Global organization of metabolic fluxes in the bacterium Escherichia coli. „Nature”. 6977 (427), s. 839–43, 2004. DOI: 10.1038/nature02289. PMID: 14985762.
↑ Reed JL, Vo TD, Schilling CH, Palsson BO. An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR). „Genome Biol.”. 9 (4), s. R54, 2003. DOI: 10.1186/gb-2003-4-9-r54. PMID: 12952533.
↑ Walsh G. Therapeutic insulins and their large-scale manufacture. „Appl Microbiol Biotechnol”. 2 (67), s. 151–9, 2005. DOI: 10.1007/s00253-004-1809-x. PMID: 15580495.
↑ Graumann K, Premstaller A. Manufacturing of recombinant therapeutic proteins in microbial systems. „Biotechnol J”. 2 (1), s. 164–86, 2006. DOI: 10.1002/biot.200500051. PMID: 16892246.
Bibliografia |
- Władysław J.H. Kunicki-Goldfinger: Życie bakterii. Warszawa: Wydawnictwo Naukowe PWN, 1998. ISBN 978-83-01-14378-7.
Linki zewnętrzne |
DSMZ Bacterial Nomenclature (ang.)
Genera of the domain of Bacteria (ang.)
Bacterial Motility and Behavior – Movies (ang.) – filmy przedstawiające różne kultury bakterii
Kontrola autorytatywna (takson):
LCCN: sh85010813
GND: 4004296-0
NDL: 00570000
BNCF: 1792
- WorldCat